接触生物信息有段日子了,自己也发表了几篇数据挖掘的文章,感觉数据挖掘很大程度上来说是在做两件事:1.比较(异同) 2.富集(特征)。
举个例子来说,如果我们对control-treatment做差异表达分析,算法会给出的差异表达基因list,按照某个统计量,比如fold change,也就是control相较于treatment的变化倍数,从小到大排序,得到一个rank list,怎么从这个list中解读出有值得研究的信息呢?也就是如何富集呢?比较常见的办法就是选取fold change(变化倍数)最大值和最小值的基因按顺序一个一个查文献,如果恰好有课题需要的蛋白,就算是中奖了,然而更多的时候我们对于差异基因两眼一抹黑,不知道它位于什么通路,是否重要。那第二种方法就是GO/KEGG了,我们可以选择foldchange>2或者<-2的基因进行功能注释,看看究竟哪些通路功能被显著富集了。但这里我觉得有一个值得注意的问题,foldchange=10的基因和foldchange=2.1的基因哪个更重要些呢?如果直接进行功能富集,这样一个重要的不同就被掩盖掉了。GSEA(Gene Set Enrichment Analysis, 基因集富集分析)就是为了解决这样的问题而产生的。
基本概念
GSEA的想法其实很简单,就是看这些差异表达的基因在一些既往的通路中的富集情况。原假设是,某个通路的所有基因,在list中是随机的分布的,假如我们能观测到某个通路的所有基因富集到list中的一端,计算其富集程度,计算其统计显著性,如果小于某个cutoff,那么我们就可以拒绝原假设,认为该通路在list中富集,并且通过富集程度的打分,如果为正,则该通路倾向于在上调的基因中富集,如果为负,则该通路倾向于在下调的基因中富集。与差异表达不同,GSEA不需要指定明确的差异基因阈值,算法会根据实际整体趋势进行分析。
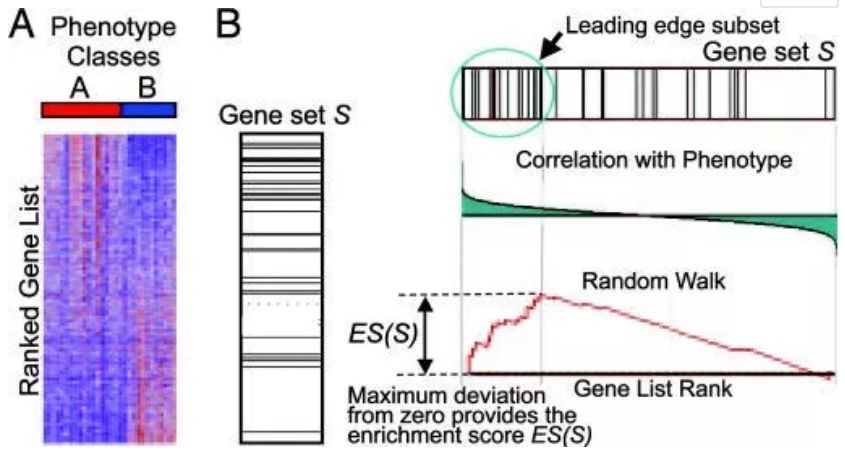
对于GSEA的结果,我们一般关注ES值(enrichment score),峰出现在前端还是后端(ES值大于0在前端,小于0在后端)以及Leading-edge subset(即对富集贡献最大的部分,领头亚集);在ES图中出现领头亚集的形状,表明这个功能基因集在某处理条件下具有更显著的生物学意义。具体的算法原理也不是很复杂,这里给大家简单介绍下。
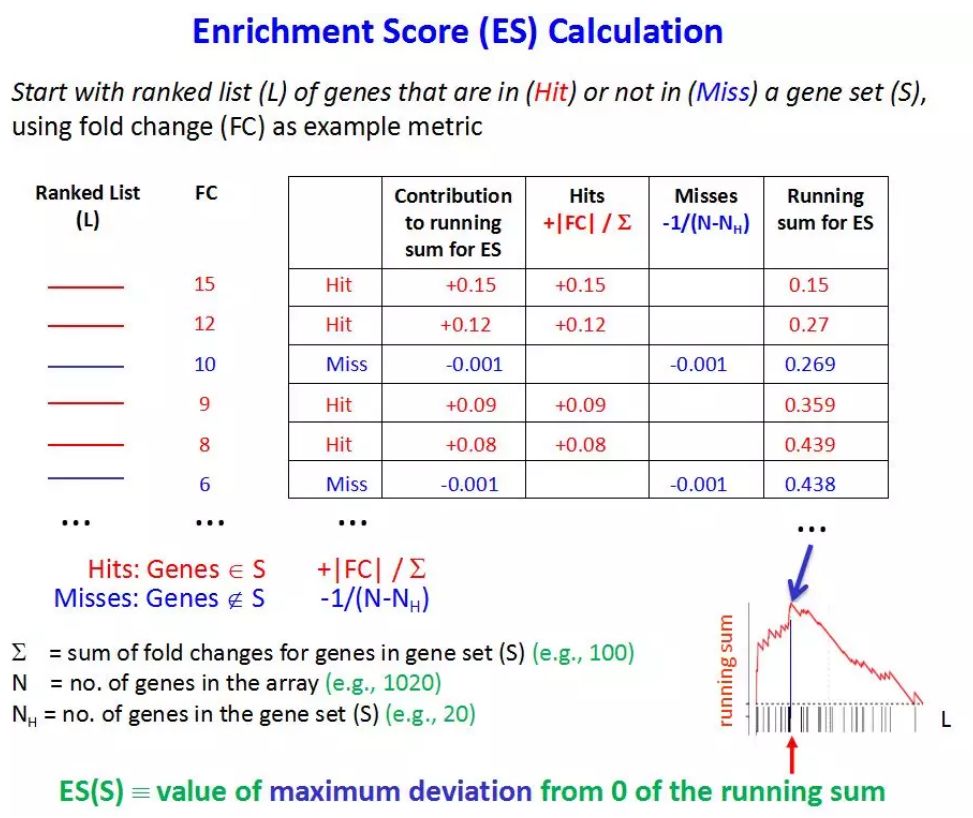
一大堆公式是不是?哈哈,看似复杂,其实很简单啦,假如某个基因属于这一通路,那么它的fold change值除以fold change总和就是它的running ES,假如它不属于这个通路,那么它的running ES就等于-1除以(基因总数-这一通路基因的个数),然后按照顺序不断累加,当达到最大值是就是这条通路的ES。
值得注意的是,官方GSEA软件默认使用signal2noise,还可以选择其他值,而今天我们的方法则采用fold change。

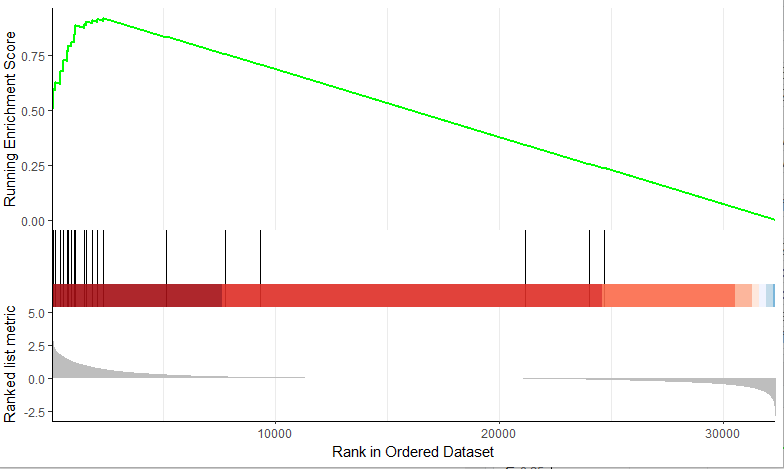
在实际操作的过程中,很多同学从软件安装都开始报错,还有就是文件格式的整理,由于GSEA需要读入特定格式的表达谱文件和表型文件,很多同学在实际操作中可能要花费很多时间来制作这两个文件。我们通过GSEA软件可以分析得到如下图:
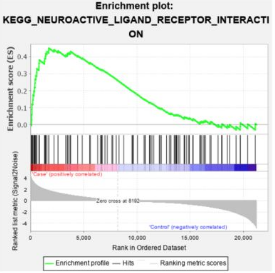
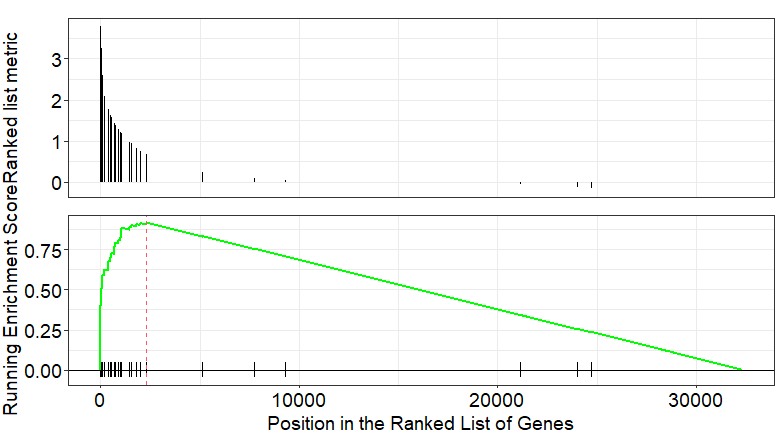
有没有什么快捷的方法,可以不要在制作各种不同格式的文件,也不要按照Java运行环境等,直接在R语言中运行GSEA呢?当然有!同时,在这个颜值取胜的时代,上面这种灰头土脸的图怎么能配得上你的CNS呢!有没有更好看的图呢?当然也有!
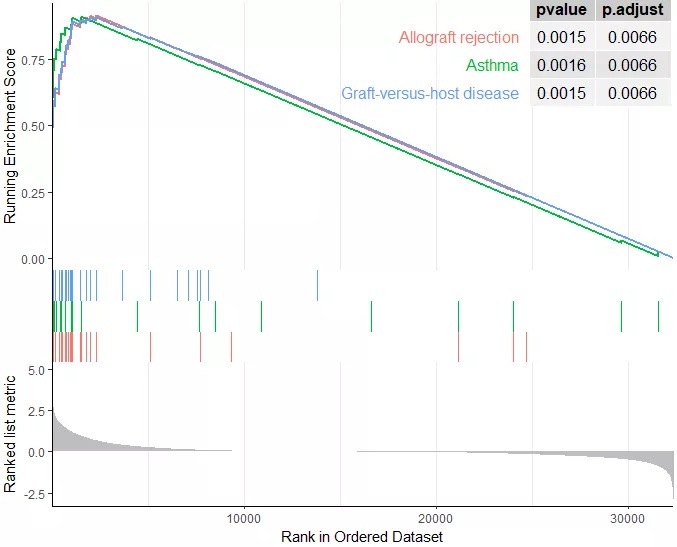
同样是GSEA,这张图有没有让人眼前一亮呢?讲完了大致原理,今天就来教给大家如何用快速做出上面的GSEA图。
实例操作
首先是导入数据,正如前面所说,我们的测试数据其实就是一个基因列表的Fold change值。好了,我们的测试数据长这样:
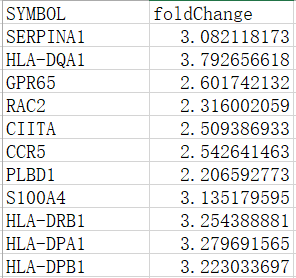
然后是ID转换,将Gene symbol转换成通用的entrze ID,最后把输入数据转换成一个包含基因fold change的数值型向量。R代码如下:
1# read in data
2df<-read.table("DEG.txt",header=T)
3head(df)
4
5# annotation
6df.id<-bitr(df$SYMBOL, fromType = "SYMBOL", toType = "ENTREZID",OrgDb = "org.Hs.eg.db")
7name.df<-merge(df,df.id,by="SYMBOL",all=F)
8head(name.df)
9
10# sort
11df.sort<-name.df[order(name.df$foldChange, decreasing = T),]
12gene = df.sort$foldChange
13names(gene) <- df.sort$ENTREZID
14head(gene)
然后就是GSEA的过程,在这里我们使用clusterProfiler包进行分析。这个R包整合了常规KEGG富集和GO富集以及KEGG、GO进行GSEA富集的函数。如发表文章,请尊重作者工作并引用文献:
“Guangchuang Yu, Li-Gen Wang, Yanyan Han, Qing-Yu He. clusterProfiler: an R package for comparing biological themes among gene clusters. OMICS: A Journal of Integrative Biology. 2012, 16(5):284-287.”
一个看似简单的工作,通过这个R包都可以快速解决,代码如下:
1# gsea
2kegg <- gseKEGG(gene, organism = "hsa",pvalueCutoff = 1)
3sortkegg<-kegg[order(abs(kegg$enrichmentScore), decreasing = T),]
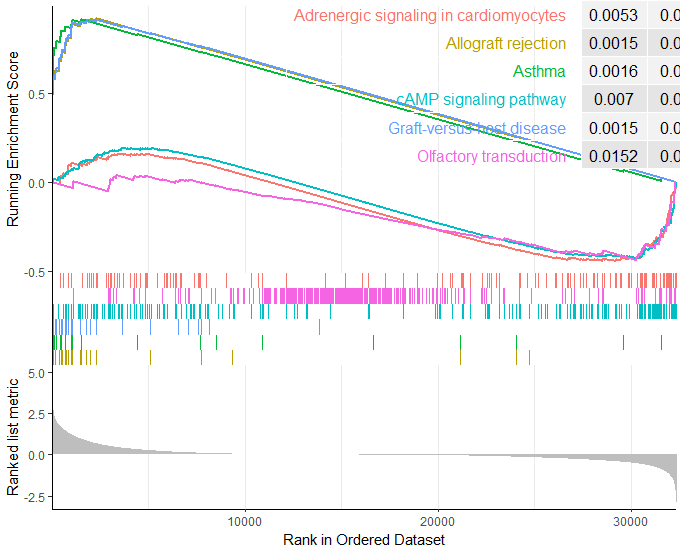
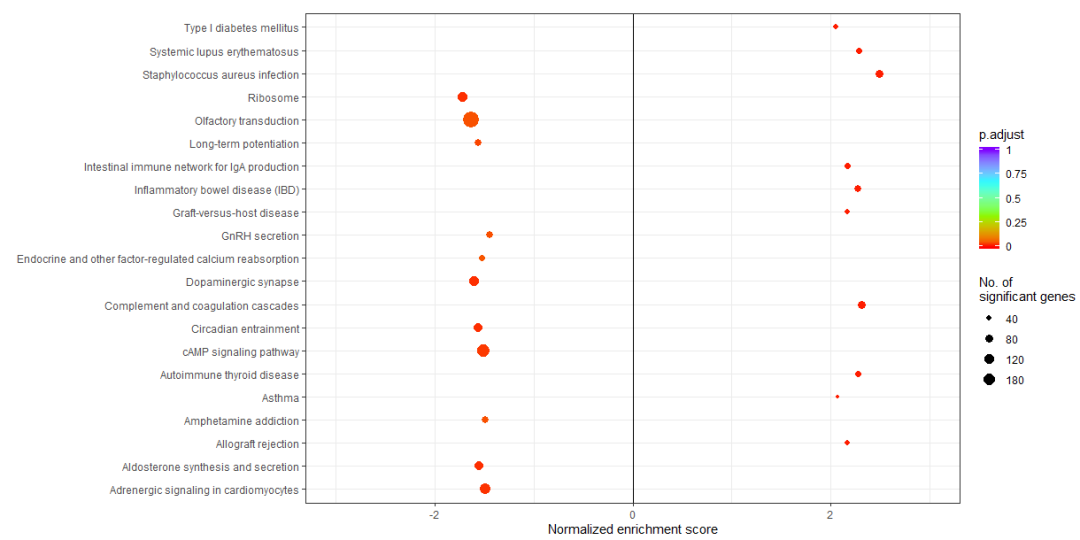
这个计算过程要耗费我们几分钟的时间,大家耐心等待一下,计算的结果我们可以把结果输出到一个csv表格当中去。最后就是画图了,如何才能绘制出高级的GSEA矢量图呢?用gseaplot()函数就可以绘制一条或者多条GSEA的曲线。如下图所示: