歌德曾说过,“一棵树上很难找到两片叶子形状完全一样”,同样的,每一个肿瘤细胞也具有各自的特征。肿瘤是恶性肿瘤细胞、免疫细胞和间质细胞的复杂混合物,通常具有较高程度的瘤内和瘤间异质性。肿瘤微环境(TME)由促肿瘤和抗肿瘤信号组成,能够调节肿瘤生长并影响肿瘤进化。
基因组和转录组的批量分析可以为肿瘤微环境内的相互作用提供有价值的见解,但批量分析提供的数据通常代表细胞的平均水平,因此往往会掩盖特定的细胞亚群和细胞状态。例如,一些潜在的“癌症干细胞”或对治疗反应至关重要的免疫细胞可能无法被检测到。而对于肿瘤免疫治疗,单细胞测序技术不仅可以为肿瘤内在或者外在的驱动机制提供独特的见解,而且可以为新免疫疗法面临的原发或者获得性耐药提供新的认知。
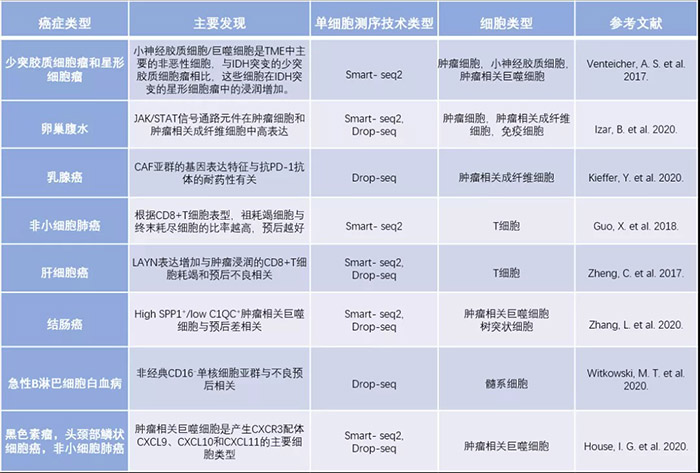
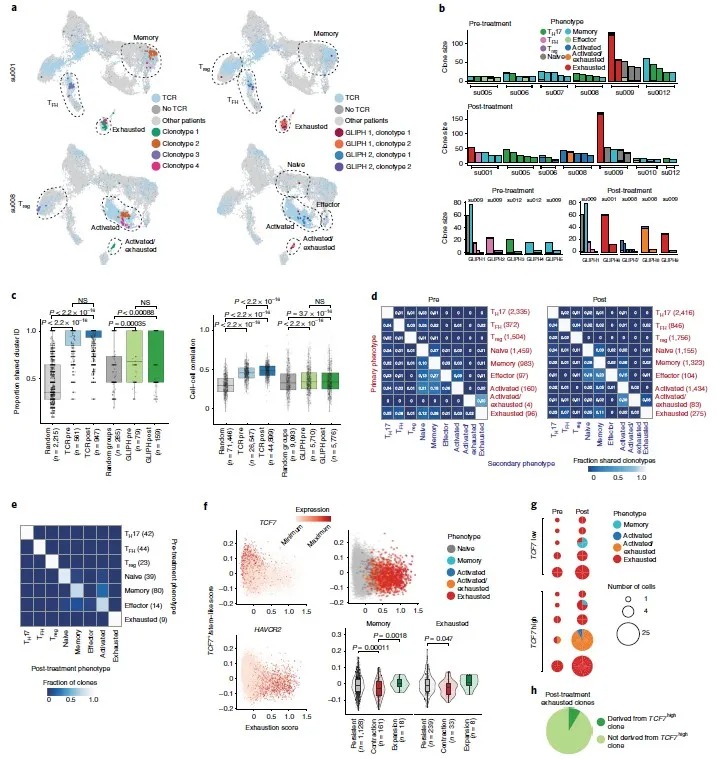
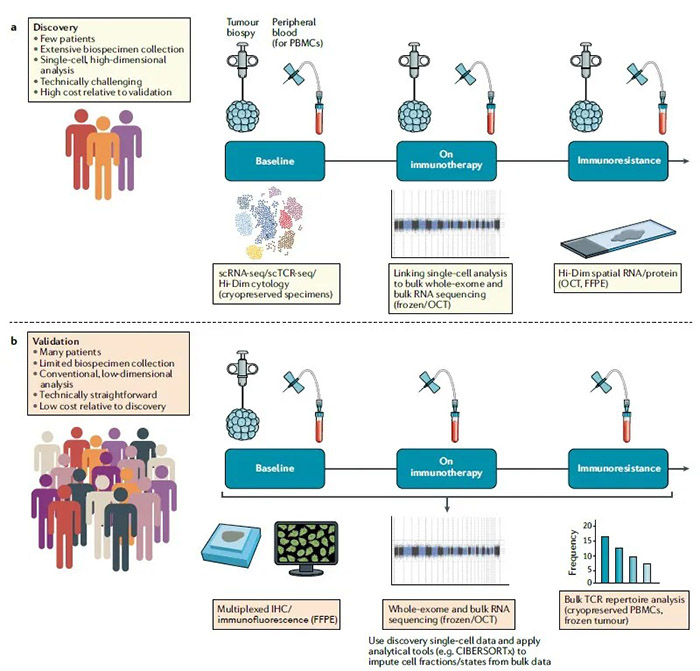
目前,单细胞测序已经回答了肿瘤免疫学中持续存在的许多问题,包括:肿瘤和免疫细胞异质性(包括抗原呈递机制表达的异质性);T细胞克隆和表型之间的关系;TME中存在的免疫检查点相互作用网络;以及肿瘤细胞和免疫细胞之间以及不同免疫细胞类型之间的空间功能关系。 在这里,我们重点关注单细胞分析如何被用来研究肿瘤细胞和它们的微环境之间的相互作用,特别是与抗癌免疫治疗反应之间的关系。然后,我们讨论了单细胞技术的未来发展方向,以及它们在推进免疫肿瘤学领域可能发挥的作用。 单细胞组学 1. 什么是单细胞转录组测序? 单细胞RNA测序(scRNA-seq)是指对单个细胞中存在的转录本进行非靶向定量的一系列技术。作为研究转录组的一种非靶向定量方法,scRNA-seq可以用来识别新的细胞类型(如肺部离子细胞,是肺部新发现的一类细胞,与囊性纤维化及其相关)(Plasschaert, et al. 2018),发现罕见细胞群,并确定细胞状态和系统发育的图谱(外周血中人树突状细胞(DC)以及单核细胞的重新分类)(Villani, A.-C. et al. 2017)。单细胞方法学工作流程,包括确定单细胞、减少高维数据、无监督聚类、系统发育建模、协调多个数据集、轨迹推断、RNA速率分析、谱系追踪和配体-受体相互作用图谱等。 肿瘤免疫学中单细胞分析的工作流程 (图片来源:Satyen H Gohil, et al. Nat Rev Clin Oncol., 2020.) 2. 如何使用单细胞测序技术分析肿瘤细胞与免疫治疗应答? 在使用scRNA-seq技术分析肿瘤细胞时,无论是在肿瘤之间还是在单个肿瘤内,肿瘤异质性都是突出的主要特征。例如,有研究者将不同肿瘤细胞(如胶质瘤、黑色素瘤、头颈部鳞状细胞癌和卵巢癌)合并在一起进行聚类分析时发现,单个细胞组的转录图谱可以按照患者来源进行区分。这样的观察结果恰恰说明了肿瘤间的高异质性,并且也证明了每一种肿瘤组织都有独特的进化轨迹。尽管存在这种异质性,但是我们可以识别具有特定转录状态的肿瘤细胞,并且这些具有特定转录状态的细胞类群在患者之间往往是相同的。到目前为止,我们已经可以根据单细胞转录组特征对肿瘤类型以及细胞类群进行区分,并且还能够识别与治疗响应或耐药相关的转录组特征。 例如,来自美国达纳-法伯癌症研究所和布罗德研究所的研究人员通过对PD-1耐药的黑色素瘤患者的样本进行单细胞RNA测序,鉴定出部分肿瘤细胞中存在一种T细胞耗竭和免疫逃逸相关的耐药性程序(Jerby- Arnon, L. et al. 2018)。当使用联合免疫治疗时(PD-1抑制剂+CDK4/6抑制剂),CDK4/6抑制剂可以抑制这类肿瘤细胞内的免疫抑制程序,并诱导其衰老,从而减少黑色素瘤的生长。而研究人员正是借助scRNA-seq这一技术手段,成功识别患者的临床特征,并建议采用新的治疗策略来克服免疫治疗耐药。 当然,除了有效的识别耐药肿瘤细胞的转录特征,全面了解免疫细胞的组成和状态对于理解当前免疫疗法的治疗效果和耐药性,以及合理设计新的免疫调节疗法和组合是至关重要的。 3.如何使用单细胞测序技术分析免疫细胞、基质细胞以及免疫治疗应答? 到目前为止,越来越多的研究者将目光集中在那些驻留于非恶性肿瘤组织中的免疫细胞上,并致力于建立详细的免疫图谱,以更有效地识别免疫微环境。 例如,Szabo, P. A. et al.(2019)通过分析来自肺、淋巴结、骨髓和血液的50,000多个静息和激活的T细胞,生成了健康人群的T细胞激活的参考图。然后,作者将公共数据库中已发表的非小细胞肺癌、结直肠癌、乳腺癌和黑色素瘤患者的肿瘤相关T细胞的scRNA-seq图谱投影到他们的T细胞激活状态图上,进而发现肿瘤中同样富集激活的CD8+T细胞,而不是功能性激活的CD4+T细胞。此外,这些激活的CD8+T细胞不仅高表达PD-1、CTLA-4等耗竭标志物,还表达与常规CD8+T细胞效应功能和持续增殖相关的基因。这些结果表明,肿瘤微环境内充斥着功能受损的CD8+T细胞。 将单细胞转录图谱扩展到一系列肿瘤类型的TME中,我们获得了关于癌症免疫应答异质性和多样性的几个有趣发现(表一)。其中一个重要的发现是,通过scRNA-seq技术对细胞进行分类,通常不会根据细胞表面蛋白的表达将其分离成传统的免疫细胞亚群,而是根据细胞的转录组状态,将其分为更贴近临床与实际情况相关的亚群,这使我们对免疫细胞在TME中的作用有了更加细微的理解。 表一:部分基于单细胞测序技术的肿瘤免疫学进展 而在免疫治疗的背景下,将这些方法应用于TME中存在的免疫和基质细胞,使人们能够阐明在免疫治疗(如免疫检查点抑制剂)期间,患者产生应答和耐药时,体内免疫和基质细胞所处的转录状态。 例如,Sade- Feldman, M. et al.(2018)等用单细胞RNA测序方法检测了来自32例接受检查点免疫治疗的黑素瘤患者的48个样本共16291个CD45+免疫细胞的转录组,并分析了影响检查点免疫治疗效果的因素。他们发现,CD8+T细胞中转录因子TCF7阳性表达可预测检查点免疫治疗的反应和较好预后。而对免疫治疗无应答的患者体内,CD8+T细胞高表达T细胞耗竭标志物(如ENTPD1、BATF、HAVCR2、PDCD1、LAG3和CTLA4)。因此,作者根据细胞的转录状态,使用双重阻断CD39和TIM3或其它检查点分子降低了肿瘤生长速率,提高了患者生存率。 总的来说,对于研究者而言,重要的是可以通过绘制各种肿瘤类型的免疫图谱来分析免疫微环境内新的细胞转录状态组合,这些状态与淋巴细胞、树突状细胞、单核细胞、巨噬细胞和成纤维细胞的免疫治疗应答相关,并且其中许多正在作为假定的生物标记物进行研究(表一)。在对肿瘤及其微环境成分的转录格局有了更好的了解后,研究人员可以在单细胞水平上研究肿瘤和免疫系统在治疗和复发过程中是如何进行协同进化的,从而为癌症治疗提供更加精确和稳定的治疗方法。 什么是单细胞T细胞受体分析? T细胞是适应性免疫系统中负责抗肿瘤免疫的主要武器,因此成为大多数FDA批准的癌症免疫疗法(如ICIS、过继细胞疗法、肽或RNA疫苗、基于细胞因子的疗法和溶瘤病毒疗法)的主要焦点。由于T细胞的这一特性,个体T细胞克隆型的分析引发了越来越多的研究兴趣。 T细胞受体(T cell receptor, TCR)是T细胞表面的特异性受体,负责识别由主要组织相容性复合体(MHC)所呈递的抗原。T细胞受体是异源二聚体,由两个不同的亚基所构成。95%的T细胞的受体由α亚基和β亚基构成,另外5%的受体由γ亚基和δ亚基构成。T细胞受体与MHC所呈递的多肽的特异性结合会引发一系列生化反应,并通过众多的辅助受体、酶和转录因子激活T细胞,促进其分裂与分化。机体淋巴细胞中起抗肿瘤作用的主要是细胞毒性T细胞(CTL),CTL表面的T细胞受体可以识别肿瘤细胞表面HLA呈递的抗原肽,攻击和破坏肿瘤细胞。 越来越多的证据表明,许多肿瘤浸润性T细胞只是“旁观者”,无法识别和攻击肿瘤细胞。因此,某些肿瘤可能看起来是被免疫效应细胞高度浸润,但由于缺乏具有抗肿瘤活性的肿瘤特异性T细胞,因此这些肿瘤仍然是“质冷”的。 scRNA-seq技术除了可以量化临床肿瘤学视角下的细胞组成和转录状态外,还能够识别成对的α和β TCR亚单位,这是确定浸润T细胞特异性的关键一步。这些信息不仅有助于解释当前免疫疗法下的耐药性(如缺乏肿瘤特异性)问题,而且有助于发现高亲和力的抗肿瘤TCR,并且这些TCR还可用于基于TCR的新型细胞工程疗法。 在此之前,人们主要利用流式细胞术和针对TCRBV亚群的单克隆抗体的组合,在蛋白质水平上进行了开拓性实验以解剖T细胞库。这种方法是定性和定量的,但受限于特定单克隆抗体的可用性,无法提供任何关于TCR互补决定区(CDR3)多样性的信息。在接下来的几年里,许多基于聚合酶链式反应(PCR)的技术被开发出来,使我们能够对单个T细胞TCR的CDR3序列进行排序,以便明确地将TCR αβ亚单位分配给肿瘤中的单个T细胞。 尽管批量T细胞测序有助于进一步了解TCR多样性,但是,以群体细胞为目标的批量测序不能确定T细胞群内特定的αβ受体链配对信息。而在单细胞水平分析TCR的价值在于:针对特定的TCR αβ链配对介导抗原特异性,可以获得与之相对应的配对信息。这对于深入了解抗原识别,靶向免疫治疗TCR的有效设计至关重要,也有助于确立T细胞群的祖先关系。 最近,Brigid S Boland, et al.(2020)利用转录组和抗原受体测序策略来鉴定患者的组织和免疫细胞特征。研究人员利用单细胞测序数据揭示了溃疡性结肠炎中组织驻留记忆T细胞(TRM)的异质性,包括炎性CD8+ TRM亚群的扩增。这种TRM亚群以及其他T细胞和B细胞亚群的鉴定为将来的功能研究提供了一个平台,有望了解这些亚群如何触发慢性粘膜损伤。 另外,联合scRNA-seq和scTCR-seq可以为分析免疫治疗过程中免疫细胞的表型和功能特征提供一种新的有效的方法。例如,对接受抗PD-1抗体治疗的晚期基底细胞癌(n=11)或鳞状细胞癌(n=4)患者的治疗前和治疗后活检样本的分析显示,治疗后激活的和长期激活的(耗竭的)CD8+TIL的数量都增加了。值得注意的是,在治疗后耗尽的CD8+TIL中观察到了最大程度的TCR克隆性(意味着多样性水平最低),并且这些克隆型中的绝大多数由抗PD-1治疗之前没有检测到的扩展的新克隆组成。治疗前存在的耗竭的TIL克隆在治疗后不会扩大,也不会转变为未耗竭的表型。对治疗后外周血标本的批量谱系分析显示,11.8%的治疗后肿瘤浸润性淋巴细胞是新的耗竭T细胞克隆型,尽管在治疗前的肿瘤浸润性淋巴细胞中未检测到这些克隆型。这些结果表明,先前存在的肿瘤特异性肿瘤浸润性淋巴细胞只有有限的再激活能力,免疫检查点抑制剂可以诱导新的T细胞克隆的涌入和扩张(克隆性替换)(Kathryn E Yost, et al. 2019.)。 肿瘤浸润T淋巴细胞的克隆动力学和表型转变 (图片来源:Kathryn E Yost, et al. Nat Med., 2019.) 总而言之,单细胞转录组技术已经可以通过T细胞受体基因配对测序来追踪单个T细胞的克隆能力,并且可以为肿瘤免疫学提供丰富的临床相关标志物数据。并且T细胞受体库分析已经成为了解健康个体和多种病理条件下T细胞生物学的基本工具,目前不仅应用于研究免疫介导性疾病的生物学,还应用于监测治疗后的免疫反应。 单细胞测序技术未来的方向是什么? scRNA-seq技术以及它的多模式技术联用都是革命性的研究工具,它们促进了我们对人类肿瘤免疫学的理解,也推动了我们对免疫疗法的应答和耐药性机制的理解。虽然免疫疗法已经改变了许多晚期恶性肿瘤的治疗方式,但大多数患者要么对这些药物没有反应,要么最终会产生耐药性。 目前,探索与其他免疫治疗药物、非免疫治疗药物或两者兼而有之的联合免疫治疗方法的临床试验已有数千项。然而,研究者及医务工作者想要设计合理的包含免疫治疗的组合以克服原发或获得性耐药性,就需要借助全面的描述各类癌症类型的肿瘤免疫微环境,并在治疗背景下详细了解有效的抗肿瘤免疫的各种决定因素。而单细胞技术能够全面分析肿瘤免疫微环境,因此特别适合研究肿瘤和免疫细胞的异质性,以及肿瘤生物学上的这种差异是如何导致治疗耐药性的。 为了有效地利用单细胞技术来提高我们对免疫治疗反应和耐药机制的理解,并帮助开发新的治疗方法,临床试验必须精心设计,以可行的方式纳入单细胞方法。目前的许多技术价格都比较昂贵,使其无法在临床上常规使用。不过,如果临床研究是与转化型科学家合作精心设计的,那么在部分患者中纳入单细胞分析是可行的,而且可以得到可应用于更大临床队列的丰富的临床数据。但是,在使用单细胞测序之前,必须认真考虑需要采集什么样的组织类型(其中包括肿瘤组织、非恶性邻近组织以及外周血PBMC)、组织保存方式(包括新鲜的、速冻的以及福尔马林固定石蜡包埋的)、以及组织最佳的采集时间(治疗前、继续治疗和治疗后),另外还要考虑如何快速处理样本材料以便立即进行单细胞分析(或冷冻保存以供以后分析)。 目前对于经费有限以及患者样本较少的研究者而言,如何从少量样本中挖掘到丰富的信息往往是具有挑战性的,但是研究者可以利用已发表的scRNA-seq数据帮助其获得更多的临床信息。此外,数据库中已有的scRNA-seq数据正在逐步建立起人类肿瘤网络图谱。 在涉及免疫疗法的临床试验中,纳入单细胞分析的技术框架 (图片来源:Satyen H Gohil, et al. Nat Rev Clin Oncol., 2020.) 单细胞RNA测序已经成为现代医学研究中的一项强大技术,这种技术能帮助科学家们检测个体细胞及其在癌症等疾病中的行为。但是,这种技术不能应用于绝大多数保存的组织样本,且价格昂贵,目前还不能作为常规临床治疗的项目。因此,挖掘高维单细胞数据来定义低维的基因和蛋白质panel,已经显示出作为预测生物标记物的前景,为临床应用提供了一条清晰的道路。反过来,通过使用生物信息学算法将批量RNA-seq数据映射到由scRNA-seq技术定义的细胞类型上,也可以获得丰富的临床数据。 2019年,斯坦福大学医学院的研究人员发明了一种名为CIBERSORTx的计算技术,可以直接分析从全组织样本或数据集中提取的单个细胞RNA。这种工具不仅能够识别细胞类型,还能识别特定环境中的状态或行为,从而发现新的作用机制,改善治疗方法(Newman, A. M. et al. 2019.)。 当然,除了单细胞测序技术以外,传统的技术手段如免疫组织化学和流式细胞术也可以提供额外的补充信息。对于血液系统恶性肿瘤和其他容易分散成单细胞的样本,含有更多荧光标记物(如20-50个)的质谱流式细胞技术和流式细胞术panels已经开始成功地用于临床试验(NCT03734198、NCT03915379和NCT04181827)。 小 结 目前,单细胞RNA测序以及它的延伸技术正日益成为发现免疫治疗应答和耐药性的重要研究工具。此外,单细胞TCR测序技术可以用来分析确定T细胞抗原特异性,并提供有关获得性免疫系统在肿瘤形成、生长和治疗中作用的关键信息。 近年来,新兴的肿瘤免疫疗法蓬勃发展,考虑到肿瘤的异质性以及患者个体免疫系统的差异,癌症治疗的未来将会是联合疗法。因此,通过单细胞测序技术获得的多维生物标记物信号对于医务工作者来说,可以为每个癌症患者做出最佳的治疗选择。另外,单细胞测序技术也可以为肿瘤免疫学提供丰富的数据与信息,为后续的研究提供支持。 注:本文编译自Gohil SH, Iorgulescu JB, Braun DA, Keskin DB, Livak KJ. Applying high-dimensional single-cell technologies to the analysis of cancer immunotherapy. Nat Rev Clin Oncol. 2020. https://www.nature.com/articles/s41571-020-00449-x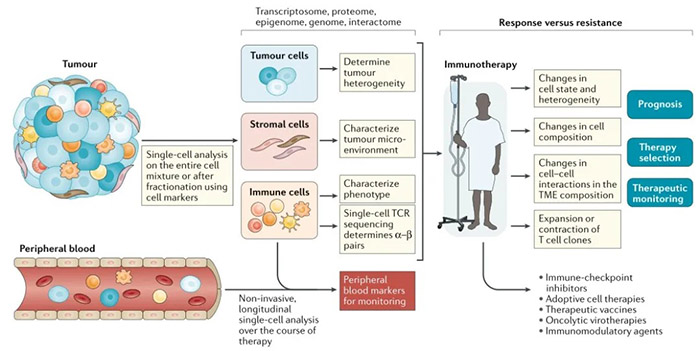
参考文献
1. Plasschaert, L. W. et al. A single- cell atlas of the airway epithelium reveals the CFTR- rich pulmonary ionocyte. Nature 560, 377–381 (2018). 2. Villani, A.-C. et al. Single- cell RNA- seq reveals new types of human blood dendritic cells, monocytes, and progenitors. Science 356, eaah4573 (2017). 3. Venteicher, A. S. et al. Decoupling genetics, lineages, and microenvironment in IDH- mutant gliomas by single- cell RNA- seq. Science 355, eaai8478 (2017). 4. Izar, B. et al. A single- cell landscape of high-grade serous ovarian cancer. Nat. Med. 26, 1271–1279 (2020). 5. Kieffer, Y. et al. Single- cell analysis reveals fibroblast clusters linked to immunotherapy resistance in cancer. Cancer Discov. 10, 1330–1351 (2020). 6. Guo, X. et al. Global characterization of T cells in non- small-cell lung cancer by single- cell sequencing. Nat. Med. 24, 978–985 (2018). 7. Zheng, C. et al. Landscape of infiltrating T cells in liver cancer revealed by single- cell sequencing. Cell 169, 1342–1356.e16 (2017). 8. Zhang, L. et al. Single- cell analyses inform mechanisms of myeloid- targeted therapies in colon cancer. Cell 181, 442–459.e29 (2020). 9. Jerby- Arnon, L. et al. A cancer cell program promotes T cell exclusion and resistance to checkpoint blockade. Cell 175, 984–997.e24 (2018). 10. Sade- Feldman, M. et al. Defining T cell states associated with response to checkpoint immunotherapy in melanoma. Cell 175, 998–1013. e20 (2018). 11. Newman, A. M. et al. Determining cell type abundance and expression from bulk tissues with digital cytometry. Nat. Biotechnol. 37, 773–782 (2019). 12. Brigid S Boland. et al. Heterogeneity and clonal relationships of adaptive immune cells in ulcerative colitis revealed by single-cell analyses. Sci Immunol. 5(50):eabb4432 (2020). 13. Kathryn E Yost. Et al. Clonal replacement of tumor-specific T cells following PD-1 blockade. Nat Med. 25(8): 1251–1259 (2019).














