RNA干扰(RNA interference, RNAi)是指在进化过程中高度保守的、由双链RNA(double-stranded RNA,dsRNA)诱发的、同源mRNA高效特异性降解的现象。它是生物进化过程中遗留下来的一种在转录后通过RNA调控基因表达的机制。
历史
1990年,Napoli和Van der Krol等在研究矮牵牛花查尔酮基因时发现了基因共抑制现象(cosuppression)。她在体外构建了控制矮牵牛花花色的基因查尔酮基因片段,连上花椰菜花叶病毒的35S启动子,连入农杆菌的T-DNA质粒,在矮牵牛花中过度表达。她原先预期,查尔酮的过度表达能加深牵牛花花色的紫色,但是结果却导致了矮牵牛花子代花色的褪色,内源性的查尔酮遭到沉默。Napoli教授把这一现象称之为内源性基因共抑制现象。
1995年,Guo等发现注射正义RNA(senseRNA)和反义RNA(antisense RNA)均能有效并特异性地抑制秀丽新小杆线虫(C. elegans)par-1基因的表达,该结果不能使用反义RNA技术的理论做出合理解释。直到1998年,Fire和Mello课题组接手了此课题。他们以秀丽新小杆线虫为模型,发现在Guo的课题中,引发线虫par-1基因沉默的是小片段的双链RNA,而不是正义单链RNA或负义单链RNA。他们之后又研究了秀丽新小杆线虫的unc-22基因,进一步阐述了双链RNA在基因沉默中的作用,并将这一现象命名为“RNA干扰”。他们的研究成果激起了其他科学家研究RNA干扰现象的浓厚兴趣,由于他们的发现揭示了分子生物学中一个全新的,具有普遍性的机制,AndrewFire, Craig C. Mello两位科学家因此在2006年获得诺贝尔奖。
原理
基因沉默分为转录水平的沉默(TGS)和转录后水平的沉默(PTGS)。TGS是指转基因在细胞核内RNA合成受到了阻止而导致基因沉默;对于部分植物来说,转基因引发的基因沉默可能是因为基因特异的甲基化而导致;PTGS则是指转基因能够在细胞核里被稳定地转录,但在细胞质里却无相应的mRNA存在这一现象。目前普遍认为RNAi、共抑制、 quelling均属于PTGS!
病毒基因、人工转入基因、转座子等外源性基因随机整合到宿主细胞基因组内,并利用宿主细胞进行转录时,常产生一些dsRNA。宿主细胞对这些dsRNA迅即产生反应,其胞质中的核酸内切酶Dicer将dsRNA切割成多个具有特定长度和结构的小片段RNA(大约21~23 bp),即siRNA。研究表明在生物体中siRNA具相似的结构特征:为长约21~23bp的双链RNA,具5’单磷酸和3’羟基末端,互补双链的3’端均有一个2~3nt的单链突出。
负责将dsRNA转化为siRNA的Dicer核酸酶,属于RNaseⅢ家族,具有两个催化结构域、一个解旋酶(helicase)结构域和一个PAZ (Piwi/Argonaute/Zwille)结构域,Dicer在催化过程中以二聚体的形式出现,其催化结构域在dsRNA上反平行排列,形成四个活性位点,但只有两侧的两个位点有内切核酸酶活性,这两个位点在相距约22bp的距离切断dsRNA,各种生物体内Dicer结构略有不同,致使siRNA长度存在微小差别。
siRNA在细胞内RNA解旋酶的作用下解链成正义链和反义链,继之由反义siRNA再与体内一些酶(包括内切酶、外切酶、解旋酶等)结合形成RNA诱导的沉默复合物(RNA-induced silencing complex,RISC)。RISC与外源性基因表达的mRNA的同源区进行特异性结合,RISC具有核酸酶的功能,在结合部位切割mRNA,导致特定基因沉默,切割位点即是与siRNA中反义链互补结合的两端。被切割后的断裂mRNA随即降解,从而诱发宿主细胞针对这些mRNA的降解反应。
在RISC中,起靶序列识别作用的是siRNA的反义链,Zamore等发现在RNAi过程中,首先产生的是RISC无活性前体,分子量250kD,当加入ATP后可形成100kD的活性复合体。由无活性前体向活性酶复合物的转换类似蛋白酶原的激活,但RISC酶复合物激活不需要共价键断裂,而要求结合于其上的siRNA双链的解开。在ATP存在时,依赖于ATP的解旋酶解开siRNA的双链并将其正义链与靶mRNA置换,mRNA取代正义链与反义链互补,然后由活化的RISC在互补区的中间,距离siRNA反义链3’末端约12bp处切断靶mRNA序列。
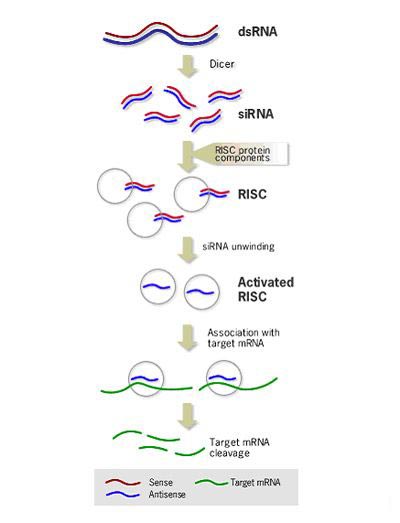
siRNA不仅能引导RISC切割同源单链mRNA,而且可作为引物与靶RNA结合并在RNA聚合酶(RNA-dependent RNA polymerase,RdRP)作用下合成更多新的dsRNA,新合成的dsRNA再由Dicer切割产生大量的次级siRNA,经过若干次的合成-切割循环,RNAi的作用不断放大,最终将靶mRNA完全降解。
siRNA 设计的原则
1.从mRNA的AUG起始密码开始,寻找“AA二连序列,并记下其3端的19个碱基序列,作为潜在的 SiRNA靶位点。有研究结果显示GC含量在30%-50%左右的SIRNA要比那些Gc含量偏高的更为有效。
2.将潜在的序列和相应的基因组数据库(人或者小鼠,大鼠等等)进行比较,排除那些和其他编码序列/ST同源的序列。例如使用BLAST。
3.选出合适的目标序列进行合成。通常一个基因需要设计多个靶序列的 SiRNA,以找到最有效的SiRNA序列。














