肿瘤微环境(Tumor microenvironment, TME)在肿瘤的发展和异质性中起着重要作用。微环境的选择力决定了肿瘤的表型特征,即适应其最佳生存环境性的细胞特性。生长在肿瘤组织和正常组织边缘的肿瘤细胞表现出促进侵袭和转移的特征,而肿瘤组织内部的细胞则通过促进新陈代谢(例如血管生成)而使细胞的增殖能力最大化。在TME中,除了恶性肿瘤细胞外,不同肿瘤类型中免疫细胞的组成和浸润程度是不同的。当更多的T细胞浸润肿瘤组织时,肿瘤组织的体积还是比较小的,患者的预后也会更好。同时,肿瘤组织的其他成分,如巨噬细胞和嗜中性粒细胞,也密切调节着肿瘤的免疫微环境。因此,不同个体对免疫疗法的敏感性具有广泛的异质性。另外,TME内的所有细胞类型都能通过细胞通讯机制彼此相互作用,增加了肿瘤发展的复杂性。了解肿瘤细胞之间的这些通信机制对于制定有效的抗癌免疫治疗策略至关重要。
为了让大家更好地了解TME研究的最新进展和研究方法,本公众号从本月起连续三个月,推出肿瘤微环境系列专题,共分为6期,每月推出2期,每期由5-6篇相关主题文章组成,并在一周内连续放送,希望能让您尽享国际上最新有关TME的硬核科研讯息和思路,为您的科研成果提供有价值的参考资料。
肿瘤异质性在癌症的进展中起着重要作用,了解单个细胞的基因表达模式尤为重要。常见的测序方法结合了数千个多个子集的细胞进行测序。因此,在肿瘤进展中起重要作用的稀有细胞克隆可能被掩盖了。在肿瘤的发展过程中不可能精确地追踪单个细胞的突变。下一代测序(NGS)方法可用于评估肿瘤细胞的异质性,以追踪异质性细胞内的基因表达或突变、评估在治疗过程中肿瘤细胞的选择性进化。单细胞RNA测序(scRNA-seq)通过以单细胞分辨率测量整个转录组并可区分肿瘤组织中的不同细胞类型,克服了传统RNA测序方法的局限性。此外,scRNA-seq使人们能够更清楚地了解促进肿瘤发生的分子机制,并揭示肿瘤进化过程中的体细胞突变。
另一方面,通过对肿瘤在不同时间点的scRNA-seq可以识别关键基因突变,以及随着时间推移细胞异质性的动态变化。同时,这一方法还能够监测肿瘤发生和发展过程中稀有细胞的突变导致的细胞功能改变,如获得侵袭和转移能力,以及免疫细胞浸润和激活及其他重要过程。最后,scRNA-seq还可以与免疫检查点疗法结合使用,以特异性检测免疫检查点的转录活性,或特异性筛选具有高转录水平的新抗原。在过去的几年中,对scRNA-seq技术的研究已经有了显着增长,为癌症的临床治疗提供了新的机遇和战略方法。随着测序技术的发展,检测的灵敏度和准确性逐渐提高,成本逐渐降低。因此,scRNA-seq已成为肿瘤研究中的重要技术工具。本公众号有关TME专题的第一期内容我们将首先关注几篇有关scRNA-seq在TME中研究的最新进展,重点是scRNA-seq在肿瘤异质性,发病机制和治疗中的应用。
那么首先开门见山,不论您是新手小白还是肿瘤研究达人,本专题的第一篇文章帮大家整理了scRNA-seq的5种技术方法以及在TME研究中的四种常见科研思路,希望能对您有帮助:
一般而言,scRNA-seq包括四个步骤:(1)分离单细胞,(2)反转录,(3)cDNA扩增,和(4)构建测序文库的。
图1显示了五种最常用的scRNA-seq方法:Tang方法,Smart-seq,基于UMI的STRTseq,CEL-seq和Drop-seq。这些方法的各有差异:Tang方法是最早的scRNA-seq技术。通过显微操作将单个细胞分离。总体测序灵敏度和准确性相对较低。在Smart-seq中,RNA被莫洛尼氏鼠白血病病毒(MMLV)逆转录。测序范围可以达到全长cDNA,具有更高的灵敏度和准确性。STRT-seq和STRT/C1-seq在Smart-seq的基础上引入UMI,并在5'端用生物素标记,可通过磁珠回收。这种测序方法提高了灵敏度和准确性,但5'端偏好性。CEL-seq通过IVT获得3′末端片段,测序灵敏度高,但3'末端偏好性强,准确性低。Drop-seq使用微流体技术将单个细胞包装为独立的液滴,从而大大提高了单个细胞的捕获能力和文库容量。它在检测大量单细胞测序样品中具有很大的优势,但是测序灵敏度低。
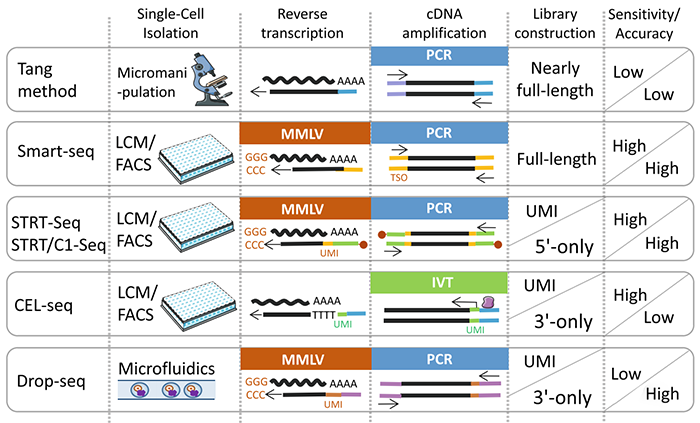
图 1
和NGS类似,scRNA-seq可用于研究肿瘤发展各个阶段的遗传和分子特征。以最严重的恶性肿瘤之一的胰腺导管腺癌(PDA)为例,该癌症类型发展的病程很长,在癌变明显表现之前可能需要数年之久。 scRNA-seq在发生胰腺上皮内瘤变(PanIN)的胰腺上皮细胞上进行,通过分析与增殖,侵袭和转移相关的基因突变,评估恶性转化的风险,从而为遏制进一步的肿瘤发展提供可能。另外,scRNA-seq可用来量化胰腺癌细胞的转录状态,并结合现有的临床数据进行PDA分型,这对于临床靶向治疗具有重要意义。Hosein等通过scRNAseq分析了PDA中从癌前病变和腺泡导管上皮化生(ADM)到恶性浸润性肿瘤的PDA进程,并分析了基因表达和细胞组成类型异质性的差异。通过发现PDA发病机理中的细胞异质,提出了靶向治疗胰腺癌的新靶标[1]。
scRNA-seq还可以提供有关单个肿瘤细胞中基因表达和单核苷酸突变的全面信息,并加深我们对原发性肿瘤转移过程的了解。Chen等通过scRNAseq鉴定了迁移细胞中差异表达的基因。他们发现了中间上皮-间质转化(EMT)状态以及迁移细胞的独特上皮和间充质亚群,从而表明乳腺癌细胞在保持上皮状态的同时迅速迁移。迁移细胞显示出氧化应激,线粒体形态和蛋白酶体调控因子的差异。他们还确定了与乳腺癌细胞迁移和预后相关的新基因,为潜在的预后生物标志物和治疗靶点,以阻止迁移性细胞的转移[2]。
除了上述在肿瘤发生研究中的应用外,scRNA-seq还可用于肿瘤异质性研究,包括肿瘤组织的细胞分型,结合恶性肿瘤微环境,遗传因素和表观遗传学的影响对恶性细胞特征性细胞的状态进行分析。Neftel等在20名成人和8名儿童胶质母细胞瘤(总共24131个细胞)中进行了scRNA-seq检测,揭示了转录和遗传的异质性,并构建了胶质母细胞瘤模型和细胞谱系。他们将胶质母细胞瘤细胞分为几个亚群,并鉴定了它们的特定细胞状态,例如(I)神经祖细胞样(NPC样),(II)少突胶质细胞样(OPC样),(III)星形细胞样(AC)和(IV)间充质 (MES)状态。此外,作者还发现了受细胞周期和微环境影响的细胞表达模式[3]。
另外,scRNA-seq的发展和应用还支持了癌症干细胞(CSC)理论。Fendler等开发了从肾透明细胞癌(ccRCC)中分离CSC的方法,并通过表达谱分析和scRNA-seq对其进行了分析。转录组和scRNA-seq表明, CSC中Wnt和Notch信号通路被激活。作者利用来自肿瘤的CSC产生了三个模型系统:非附着球培养,3D类器官和PDX肿瘤,克服了单个模型系统所带来的局限性。他们在不同阶段用针对Wnt和Notch的小分子抑制剂治疗了每个模型。异种移植治疗进一步提供证据,表明特定的患者群体可以从抑制Wnt和Notch治疗中受益。此外,作者还通过scRNA-seq发现了一个小的CSC亚组,并确定了CSC的异质性程度。以上实验结果为肿瘤耐药和CSC治疗的临床应用提供了新思路[4]。
尽管如此,scRNA-seq在检测体细胞突变和肿瘤异质性方面也有局限性。缺点包括各种测序方法中的3'或5'偏好性,低丰度转录本的敏感性低,scRNA-seq数据中的噪声水平高,单细胞水平的测序深度不足等。但是,办法总比问题多,科学家已提出解决这些问题的新方法。例如,Anna S Nam等开发了转录组基因分型(GoT)技术,将基因分型与基于高通量液滴的scRNAseq整合在一起。该技术可以同时捕获原发癌标本中成千上万个单细胞的体细胞基因型和转录组样本[5]。
肿瘤异质性包括很多方面,其中免疫异质性是重要的方面,这与肿瘤的耐药性和免疫治疗有关。如图2所示,scRNA-seq证实肿瘤组织包含复杂的免疫成分,包括先天性免疫细胞,例如树突状细胞(DC),未成熟树突状细胞(iDC),活化的树突状细胞(aDC),嗜酸性粒细胞和嗜中性粒细胞,肥大细胞 ,巨噬细胞,自然杀伤细胞(NK;NKCD56暗细胞,NKCD56亮细胞);适应性免疫细胞,例如T辅助细胞Th1和Th2,调节性T细胞(Treg),CD8 + T细胞,中央记忆T细胞(Tcm),记忆效应T细胞(Tem),T滤泡辅助细胞(Tfh)和γδ T细胞等。
凭借更高的检测精度,scRNA-seq可用于准确分类肿瘤细胞,尤其是识别免疫细胞的不同类型,包括辅助性T细胞,细胞毒性T细胞和其他T细胞(图2)。Zhang等获得了来自结肠癌患者的11138个T细胞,并系统地研究了细胞的组织分布,克隆扩增,迁移以及发育转变或分化。对不同T细胞的高表达基因和特异性T细胞受体(TCR)序列进行了分析,进一步对患者的T细胞进行分类,并描绘出该细胞在肿瘤内的免疫状况[6](图2)。因此,scRNA-seq能够分析不同肿瘤浸润免疫细胞的特定特征,从而设计靶向治疗和改良的免疫疗法,以实现更好、更具有转化价值的临床治疗效果。
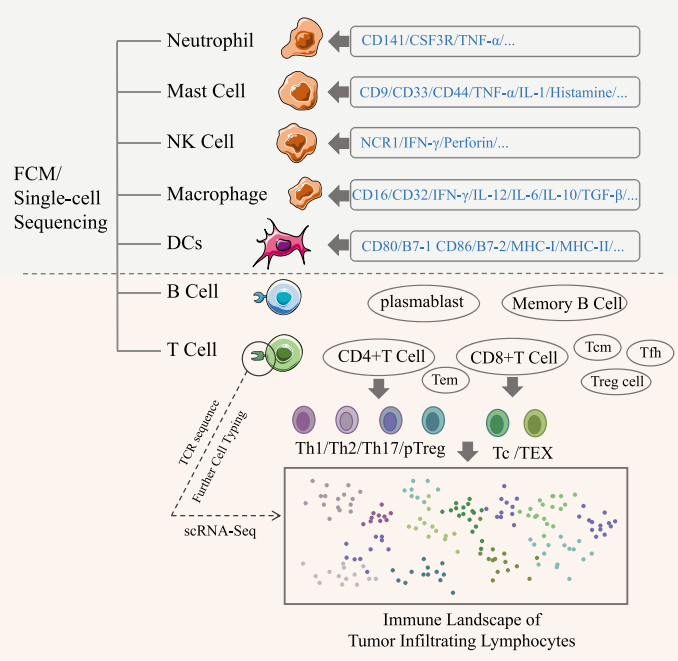
图 2
免疫监视是免疫系统最基本的功能之一,它可以通过识别、杀死和消除体内的突变细胞来防止肿瘤的发生。但是,科学技术的最新进展表明,免疫系统不仅可以保护宿主抵抗肿瘤的发展,而且还可以通过选择免疫原性低的肿瘤细胞来促进肿瘤的生长。为了更好地描述免疫系统在肿瘤发生和发展中的作用,由此提出了“免疫编辑”的概念。通过肿瘤细胞、免疫细胞和肿瘤微环境之间的持续相互作用,肿瘤的发展分为三个连续的阶段:消除、平衡和逃逸。在逃逸阶段,对遗传变异和免疫攻击不敏感的存活肿瘤细胞开始以不受控制的方式增殖,从而导致临床上可观察到的恶性肿瘤。如果这个阶段不受控制,它将最终导致宿主的死亡。
scRNA-seq可用于分析肿瘤组织中免疫相关基因的异质性,通过设计针对多种肿瘤抗原的联合治疗策略,可提高免疫治疗肿瘤的临床疗效(图3)。Noemi等为CTLA-4和PD-1治疗不敏感的滤泡性淋巴瘤(FL)患者构建了一个免疫检查点的基因网络。通过FL和扁桃体样本的scRNA-seq,选择了11个免疫检查点,包括LAG3,HAVCR2(TIM3),TIGIT,CD27,CD40LG,ICOS,TNFRSF9(4-1BB),TNFRSF18(GITR)和TNFRSF4(OX40R)。通过对与这11个免疫检查点共转录和共表达的基因的分析,作者发现了免疫调节过程中的新调控基因,并在侵入性T细胞谱系中构建了免疫检查点的基因网络,从而可以进一步理解肿瘤免疫逃逸的复杂机制,为免疫治疗的进一步发展提供了思路[7]。
对于那些对CTLA-4和PD-1/PD-L1治疗不敏感的肿瘤患者,可使用scRNA-seq分析特定的肿瘤组织和肿瘤细胞并发现其耐药机制(图3)。Sangeeta Goswami等分析了接受免疫检查点治疗的94位患有五种不同癌症类型的患者,其中包括对免疫检查点治疗不敏感的患者。他们在胶质母细胞瘤患者的样本中鉴定出一组特定的CD73hi巨噬细胞,这些患者进行了抗PD-1治疗并利用scRNA-seq分析所分离的细胞。结果发现,免疫抑制基因在CD73hi细胞中的表达增强,CD73hi细胞则减少了肿瘤组织中的T细胞浸润,并降低了患者对免疫疗法的敏感性。因此,作者提出了针对CD73的联合治疗策略以及PD-1和CTLA-4的双重阻断,这一疗法可能具有很高的临床价值[8]。

图 3
scRNA-seq可用于表征肿瘤相关细胞类型的丰度和功能状态,检测细胞之间的通讯,分析不同组织细胞间相互作用的关系,以及研究肿瘤微环境的病理生理特征,从而预测肿瘤的发展和预后。这一技术还可为单细胞基因组学在靶向治疗和免疫治疗中的应用提供新思路。
Zhou等系统地比较了通过scRNA-seq从黑素瘤分离的七种细胞类型(黑素瘤细胞,T细胞,B细胞,巨噬细胞,NK细胞,CAF细胞和内皮细胞)中2558个配体-受体对的表达模式。基于特定的配体-受体连接,建立了不同细胞类型之间的相互作用网络。该研究检测到大量细胞间通讯信号,例如生长因子、趋化因子和基质蛋白,它们与肿瘤的增殖、转移、细胞粘附、血管生成和免疫调节过程密切相关,从而揭示了肿瘤细胞间相互作用网络具有重要意义,可用于研究肿瘤的发展和预后[9]。
参考文献
[1] Hosein, A.N., et al., Cellular heterogeneity during mouse pancreatic ductal adenocarcinoma progression at single-cell resolution. JCI Insight, 2019. 5. [2] Chen, Y.C., et al., Single-cell RNA-sequencing of migratory breast cancer cells: discovering genes associated with cancer metastasis. Analyst, 2019. 144(24): p. 7296-7309. [3] Neftel, C., et al., An Integrative Model of Cellular States, Plasticity, and Genetics for Glioblastoma. Cell, 2019. 178(4): p. 835-849 e21. [4] Clark, S.J., et al., scNMT-seq enables joint profiling of chromatin accessibility DNA methylation and transcription in single cells. Nat Commun, 2018. 9(1): p. 781. [5] Nam, A.S., et al., Somatic mutations and cell identity linked by Genotyping of Transcriptomes. Nature, 2019. 571(7765): p. 355-360. [6] Zhang, L., et al., Lineage tracking reveals dynamic relationships of T cells in colorectal cancer. Nature, 2018. 564(7735): p. 268-272. [7] Andor, N., et al., Single-cell RNA-Seq of follicular lymphoma reveals malignant B-cell types and coexpression of T-cell immune checkpoints. Blood, 2019. 133(10): p. 1119-1129. [8] Goswami, S., et al., Immune profiling of human tumors identifies CD73 as a combinatorial target in glioblastoma. Nat Med, 2020. 26(1): p. 39-46. [9] Zhou, J.X., et al., Extracting Intercellular Signaling Network of Cancer Tissues using Ligand-Receptor Expression Patterns from Whole-tumor and Single-cell Transcriptomes. Sci Rep, 2017. 7(1): p. 8815.














