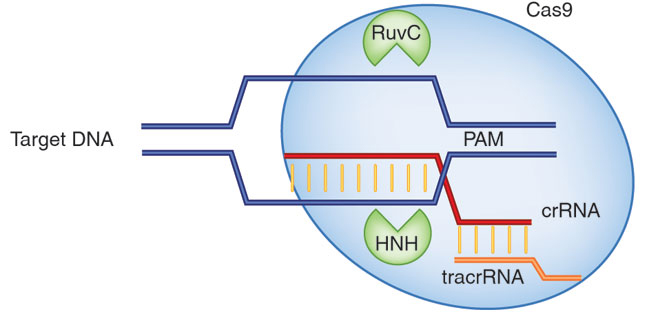
CRISPR/Cas9 是一种细菌抵抗噬菌体入侵的免疫机制,在该机制中 Cas9 蛋白含有两个核酸酶结构域,可以分别切割目标 DNA 的两条链,而 CRISPR 则是一段引导 RNA(gRNA) 负责引导 Cas9 蛋白至目的基因位置处进行切割,从而对目的基因进行编辑。
常用的 Cas9 蛋白来源于 Streptococcus pyogenes 菌株,由于该蛋白的 PAM 识别序列仅仅只有 2 个碱基(GG),因此几乎可以在所有的基因中都能找到大量的靶点,因此得到广泛的应用;并且 Cas9 蛋白在几乎所有的生物和细胞中都有活性,包括植物和动物;
saCas9 是目前自然界中发现的 II 型 CRISPR-Cas 系统中体积较为小巧的 Cas9 蛋白,由 1053 个氨基酸组成。因此,该蛋白便于体内递送,可在多种生物中进行基因组编辑。研究发现,saCas9 结合和切割靶向 DNA 分别需要 6bp 和 18bp 的 PAM 近端 DNA 与 gRNA 互补。这些酶活性是由 DNA 蛋白间两个稳定的互作位点介导的,其中之一位于距 PAM 约 6bp 处,另一个在原始间隔区域内。
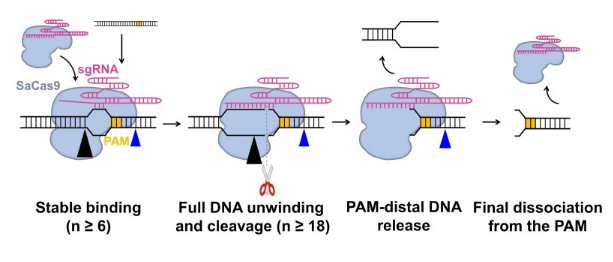
saCas9 结合、解旋、切割以及释放靶向 DNA 的动态过程示意图(DOI:10.15252/embr.202050184)
因此对于 Cas9 和 saCas9 的选用,需要根据具体实验的需求进行选择,简单的说就是,一般 Cas9 蛋白的基因敲除效率更高,并且靶点的选择性更多。而 saCas9 蛋白的特异性更高,但相对的靶点的选择性也更少。
CRISPR是指有效编辑有机体内部分基因的技术。目前根据不同的Cas核酸酶来分类一共有三种不同的系统。其中二型的比较简单,是以Cas9核酸酶以及向导RNA(gRNA)为核心组成。
如果要使用CRISPR/Cas9系统对基因组进行编辑,第一步也是最重要的一步,就是要设计gRNA。下面这篇文章就为大家汇总一下gRNA设计常用的方法:
一、首先着重介绍几个在线设计gRNA的工具
着重推荐Lei Stanley Qi Lab的http://crispr-era.stanford.edu/index.jsp
这个在线站点功能非常丰富,可以选择基因编辑工具的其他用途(激活,抑制基因表达等)(图1-1)。下一步就是选择物种(图1-2)。但只有常见的9种。再下一步就是直接输入基因的名称(或序列)即可(图1-3)。
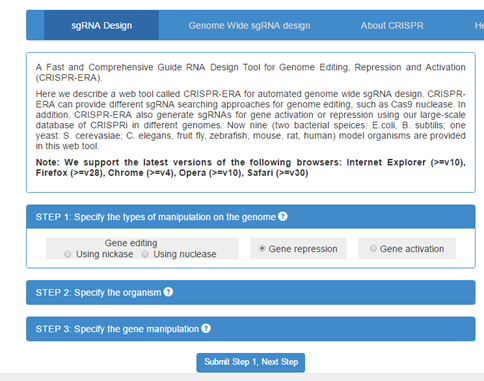
图1-1 选择编辑类型
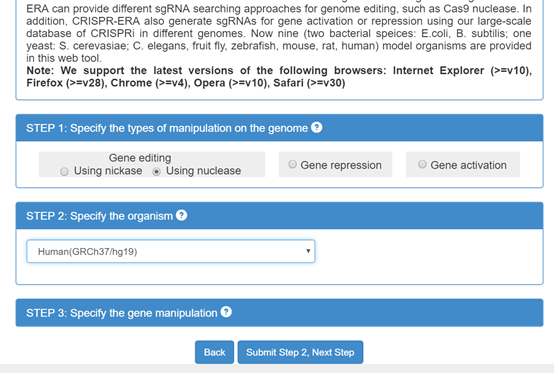
图1-2 选择物种
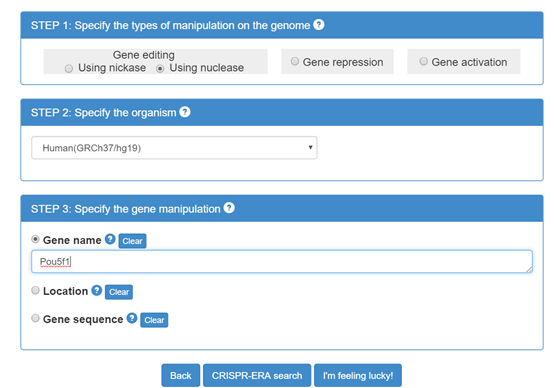
图1-3 选择基因名称
但是出来的sgRNA位置有一些并不总在ATG的下游(图2所示,1,2,6,9,12,16,22几条sgRNA位于ATG的上游)。所以根据位置,对于做基因的敲除(KO)而言,强烈建议选择ATG下游通常100 aa以内范围内的sgRNA来用(比如图3中的3,5,7,10等较下游的);而且,一定要位于CCDS区域内(CCDS是consensus CDS,即公共的CDS区域,是针对很多个转录本[isoforms]定义,图2中路色条框即是。这个很重要!!!)。
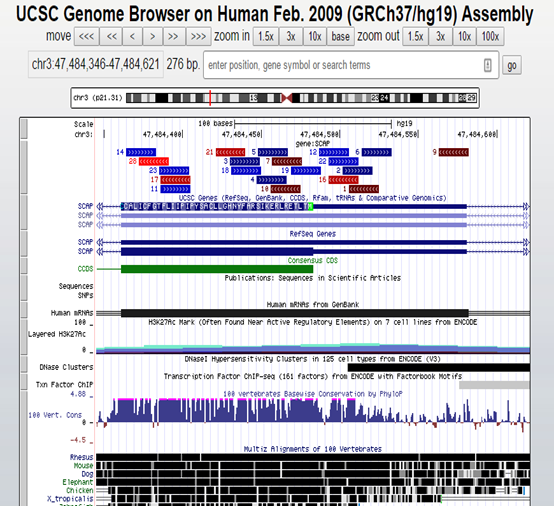
图2
● Feng Zhang lab:http://crispr.mit.edu/
这是最早的一个设计站点。包括常见的16个物种。选中物种,把你要设计的区段序列扔到下面的框里运行就行。。但前提是你自己已经选好了位置(图3)
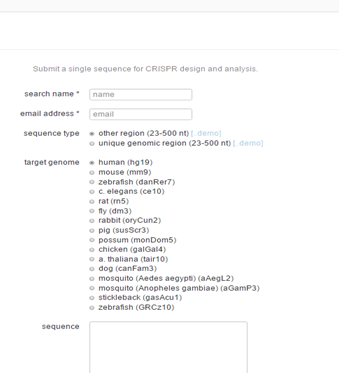
图3
还有其他,如omictools下面的Cas-Designer
http://www.rgenome.net/cas-designer/
这个站点内容更加丰富,比如物种覆盖式最多的,包括植物,昆虫等等(图4左)。这个站点也是输入目的基因的序列定制的,不同的是可以有较大的自由度选择位置预测。
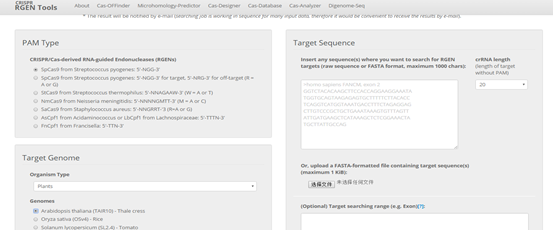
图4
还有一些商业公司的,比如Thermo下面的(图5):
https://apps.thermofisher.com/crispr/index.html#/search
物种较少,商业性目的特别强,了解下即可。
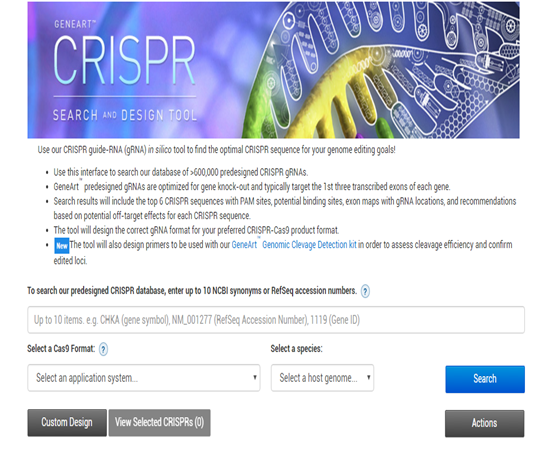
图5
其实你只要google一下会发现特别多的设计工具,感兴趣的可以自行探索。
图6
二、手动版设计方法—好用不贵
手动版本虽然费事,但有时候却会事半功倍,节约后续鉴定单克隆的成本。我们举例来说:

图7
序列中PAM和sgRNA的序列放大如下(前提是sgRNA位于CCDS区域,而且特异性要保证):

图8
也即我常说的,让Cas9切割位置(确定的)跨过一个酶切位点(比如sfcI)(在前两篇文章中都有所提及)。这样,在Cas9成功编辑靶序列产生indels的时候,酶切位点一定会遭到破坏。这为后续的单克隆鉴定提供了便利。请看示意图(图9):
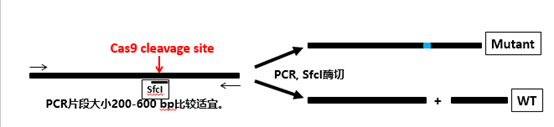
图9
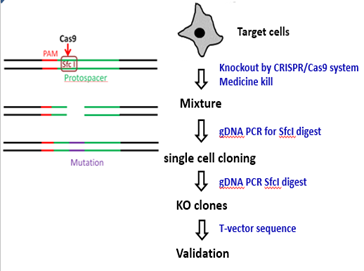
注意一点:PCR引物设计的时候确保PCR片段内部不要出现第二个SfcI的酶切位点。这样后面酶切的时候结果是唯一的。实验流程和鉴定结果应该是这样的(图10):
↓
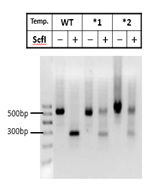
图10
对于WT,酶切是充分的,不会残留。而对于成功编辑的两个mixture:*1和*2,由于产生了indels,部分sfcI的位点造到破坏,sfcI切不动的。对于成功编辑的单克隆而言,应该是一点都切不动。后续鉴定只要这样的克隆去测序即可(图11,红色编号为阳性克隆)。
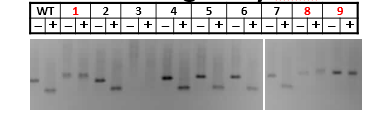
图11
TA克隆结果(图12):

图12
三、其他Cas9,比如Cpf1和saCas9的设计
因为以上介绍的站点没用涉及Cpf1和saCas9,特别是saCas9(非常适合AAV介导的载体基因编辑):http://crispr.cos.uni-heidelberg.de/index.html 这个网站兼容几乎所有类型的Cas9设计。
打开页面,输入要编辑区域的基因组序列,然后在PAM类型里选择你需要的Cas9类型,比如spCas9,saCas9以及cpf1等。其他条件默认设置即可,然后点击sunmit即可。非常傻瓜式的操作(图13,14)
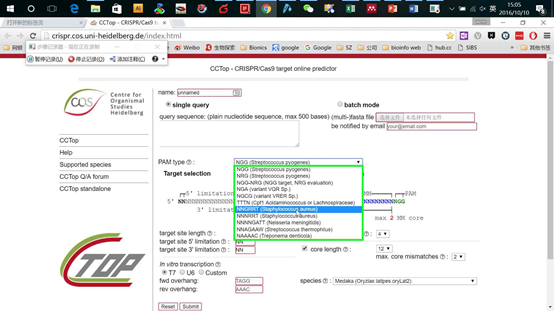
图13
在Cas9成功编辑靶序列产生indels的时候,酶切位点一定会遭到破坏
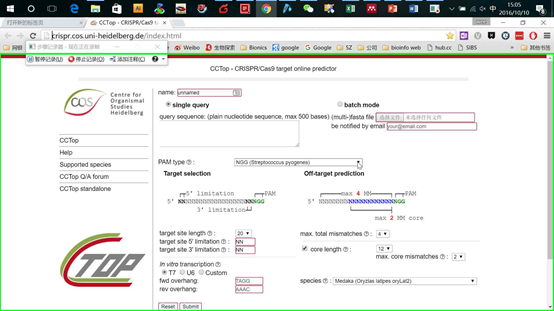
图14














