日期:2020年8月11日
杂志:PNAS
原文标题: DNA methylation enables transposable element-driven genome expansion
原文链接:https://www.pnas.org/content/117/32/19359.long
来源:美国University of Pennsylvania 和 Van Andel Institute
摘要
本文根据53种生物的全基因组数据,发现它们基因组大小的实质性差异很大程度上可以通过其中转座因子(TEs)的比例来解释。 TE与宿主共存的主要原因是CpG甲基化抑制了它们的转录。 因此,基因组扩增在很大程度上取决于DNA甲基转移酶的作用,而DNA甲基转移酶与TE大致同时进化。 CpG甲基化的长期结果是TE和宿主DNA中C-T转换突变的增加,这导致CpG二核苷酸在进化时间内的比例降低。 TE在宿主基因组中的存活还提供了额外的DNA,可将其重新用于宿主调节功能。
内容
多细胞真核基因组显示出巨大的差异。这种变化的很大一部分是由于转座因子(TEs)的存在。它们对细胞的DNA量起重要作用,并有可能参与宿主基因的控制。作者认为,通过DNA中的C-磷酸-G(CpG)二核苷酸甲基化抑制其活性对于它们在宿主基因组中的长期适应以及因此对其扩展至关重要。胞嘧啶甲基化的必然结果是通过脱氨基作用导致C到T转换突变的增加,这会导致CpG丢失。 TE经常需要胞嘧啶脱氨才能发挥宿主基因组中的调节功能。我们对53种生物的全基因组序列进行的研究显示,基因组大小与所含TEs百分比之间呈正相关,而在大小与CpG观察/预期(O / E)比之间呈负相关。 TE和宿主DNA均如此。很少在启动子和转录起始位点发现TEs,但在增强子中发现更多,特别是在它们积累了C-to-T和其他突变之后。因此,TE DNA的甲基化允许基因组扩展,也为基于TE的调控位点带来了新的基因控制机会。
真核基因组所含的DNA比其所含蛋白质编码和非编码基因所需的DNA多得多,并且它们的大小差异高达64,000倍。 尽管这些大小差异的功能意义仍然是未知的,但很多变异性可以通过存在重复的DNA来解释,尤其是多年前被Barbara McClintock鉴定出的可转座元件(TEs)。 例如,人类基因组具有三类主要的TE,它们共同构成人类DNA的45%以上:长散布的核元件(LINE),短散布的核元件(SINE)和内源性逆转录病毒(ERV)。 这些元素在进化过程中已插入自身并在波浪中转入真核种系,并具有改变宿主生物体基因控制的潜力。
三十年前,Bestor提出,DNA胞嘧啶甲基化的重要功能是沉默TEs的表达。考虑到这些元素异位表达的潜在致命作用,甲基化将使TE与宿主以某种宿主-寄生虫关系共存。一个重要的附加假设是原核DNA甲基转移酶通过保护宿主免受外源DNA整合而开始,但演变为允许外源DNA在宿主基因组中共存的酶(图1)。从原核DNA甲基转移酶的相对狭窄和稀有的序列特异性到识别简单且频繁的C-磷酸-G(CpG)二核苷酸的真核酶的过渡,使得TE可以在宿主体内适应。 Bird等人(8)使用甲基化敏感的限制性内切酶,随后发现无脊椎动物的CpG甲基化区域非常少或高度分隔,而脊椎动物具有基因间的修饰模式,且分布更为广泛(图1)。
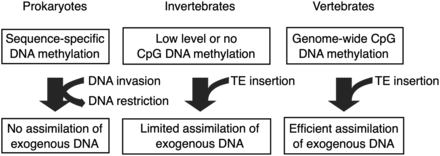
图1
该模型说明了DNA甲基化在处理外源DNA中的不同作用。 原核生物中的DNA甲基化是其宿主防御的限制/修饰系统的一部分。 由于DNA CpG甲基化的普遍性较低,无脊椎动物可以在有限的程度上容纳TE DNA。脊椎动物,尤其是哺乳动物,在基因组规模上具有广泛的CpG甲基化,并且可以耐受高水平的TE。
这些有见地的观察是在全基因组测序出现之前进行的,并未完全理解胞嘧啶甲基化本质上具有强烈的诱变性。 这是由于在甲基化位点(例如CpGs)的C到T转换突变的发生率大大提高。 反过来,这会导致具有CpG甲基化的生物体DNA中CpGs的观测/预期(O / E)比(CpG二核苷酸损失的一种度量;材料和方法)大大降低。 例如,人类DNA的CpG O / E比约为0.25,甲基化的CpG位点在种系中的半衰期约为3500万年。 通过测量该二核苷酸在现代物种中的代表性不足,我们可以推断出进化时期CpG DNA甲基化的普遍性。
通过检查53种生物的完整DNA序列,可以证实Bestor原始假设的有效性,并且发现了一些概念,即TE的整合不仅导致TE DNA的基因组扩展和甲基化,而且导致TE DNA的甲基化。 侧翼宿主DNA。 虽然扩展的进化驱动力仍是未知的,但是基因组大小和CpG代表性不足之间存在明显的相关性,这表明DNA甲基化导致DNA质量的大幅增加。 我们还证实了较早的建议,即TE可以促进新的顺式调控DNA元件的形成,而这些元件实际上是在活细胞中与转录因子结合的。 但是,一般而言,与非TE DNA产生的结合事件相比,这种贡献导致结合事件的数量适中,并且通常要求TE经历了C-to-T和其他突变形式的进化改变。
基因组大小和CpG O / E比值呈负相关。
作者使用无脊椎动物和脊椎动物的全基因组DNA测序数据来评估CpG O / E比率,基因组大小和TEs占据的基因组百分比(图2A)。对于所检查的53种生物,图2B显示了基因组大小与TE含量之间的正线性关系,图2C显示了CpG O / E比与基因组大小之间的逆关系。具有较大基因组的物种具有更高水平的基因间DNA甲基化水平,这主要归因于TE的甲基化(图2D)。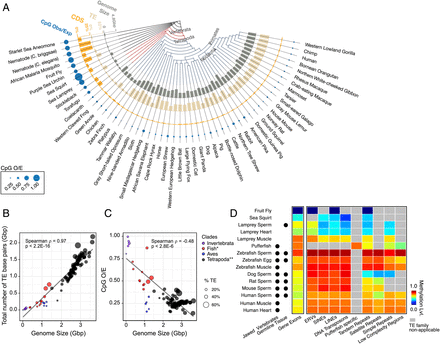
图2
从进化上讲,古老的TE在增强子上找到,但在转录起始位点上却找不到。
接下来,我们确定了TE在转录起始位点(TSS)和增强子方面的分布(图4)。 较早的研究将启动子视为一个广阔的区域,发现其中25%的区域带有TEs。 在确定了确切的TSS位置(Ensembl版本87)之后,我们发现所有三类TEs(无论它们的进化年龄如何)实际上都被强力排除在TSS之外,并且它们的频率随着与TSS距离的增加而增加(图4A)。
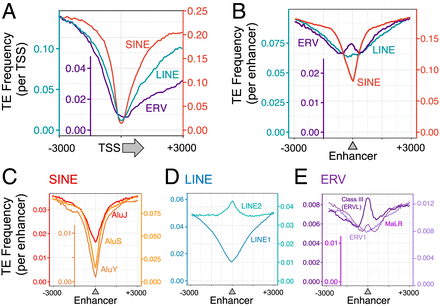
图4
宿主DNA比TE衍生DNA更容易存在TFBS
总的来说,我们发现TEs最常见于内含子和基因间区域,约占人类总DNA的45%。 TFBS的已知基序几乎在TE和非TE DNA中平均分布。 但是,与基因间DNA相比,内含子中SINE和LINE中结合的(因而具有潜在功能的)TFBS频率更高。 另一方面,ERVs中的TF结合在基因间区域更为常见。在校正到最接近的TSS的距离后,在非TE序列中TF结合比TE序列更普遍的结论
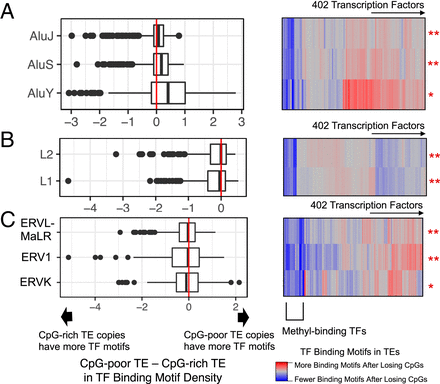
图5
总结
全基因组DNA甲基化的出现对基因组如何进化产生了深远的影响。 DNA甲基化将是理解增量进化如何被TEs和宿主DNA复杂地相互作用,共同进化并有助于大大扩展的基因组中的监管创新的系统取代的关键。














