tRFTar[http://www.rnanut.net/tRFTar/#]是一个基于CLIP-seq来预测tRFs有关的靶基因数据库。在这个数据库当中,作者一共鉴定出12103个tRFs和5688个靶基因。
背景数据集介绍
由于目前对于tRFs的研究没有像普通基因那样有很权威的类似gene、ensemble这样的数据库把基因序列注释好。所以在进行测序数据分析的时候,使用不同的tRFs序列数据库可能比对到的tRFs的结果就不一样。在tRFTars。作者首先使用的是MINTBase V2.0[https://cm.jefferson.edu/MINTbase/]进行测序数据的比对。
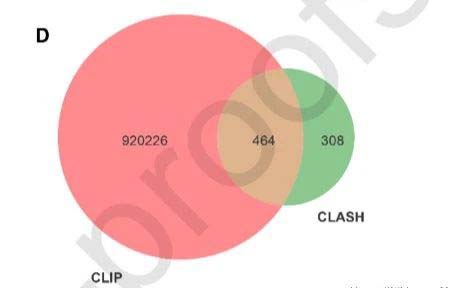
在昨天的综述当中,我们介绍了xtRFs主要发挥作用也是通过AGO蛋白发挥作用的。所以作者通过SRA数据当中的AGO CLIP-seq数据以及CLASH的数据来交叉分析预测tRFs主要是和哪些基因进行结合。同时为了更好的了解tRFs表达和基因之间的关系。作者还使用了TCGA的数据库对其进行了共表达分析。
数据库操作
这个数据库一共提供了三种不同的功能:1) 检索功能:筛选目标tRFs和目标mRNA是否存在相互作用关系;2) 富集分析:对目标tRFs进行富集分析研究; 3) 浏览功能和下载功能
检索功能
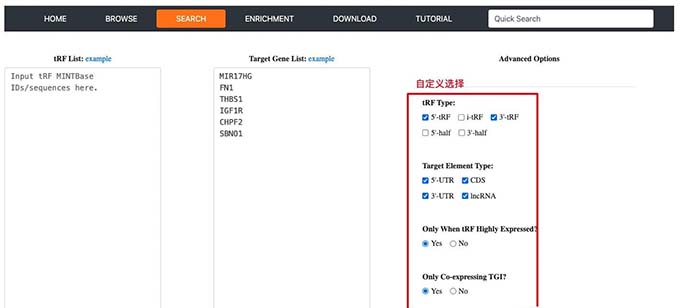
如果我们有目标基因,但是想要预测这些基因是否收到tRFs的调控。就可以使用这个检索功能。
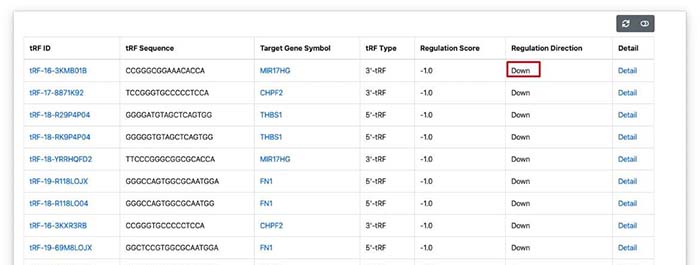
在输入完之后,就可以获得可能调控这个基因的tRFs。在结果当中,我们可以具体哪些tRFs可以调控目标基因。同时可以可以看到具体的调控方向是高表达还是低表达了。
同时,如果我们点击Detail就可以获得关于这目标tRFs以及目标基因的具体信息。

富集分析
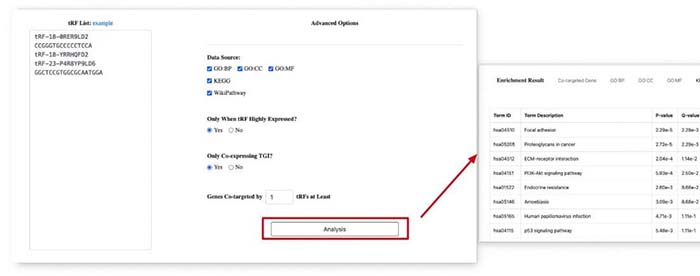
如果想要从tRFs研究入手的话,可以通过富集分析的方式来来了解具体tRFs的功能。进而来选择目标靶基因进行研究。在tRFTar当中。提供了GO、KEGG、WikiPatyway三个背景数据库用来分析。经过分析之后,就可以获得相对应的富集分析结果了。
数据下载
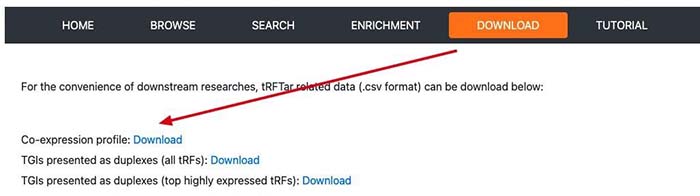
除了以上基于数据库的分析。数据库也提供了原始的数据的下载。方便我们使用。
tRFTars
除了上面介绍的tRFTar。和这个数据库一起发表的还有一个tRFTars[http://trftars.cmuzhenninglab.org:3838/tar/]。同样的数据库,在功能上和上一个比起来就稍微少了一些了
背景数据集介绍
tRFTars在tRFs注释上使用的是tRFdb[http://genome.bioch.virginia.edu/trfdb/]数据库的资料。相较于上面的MINTBase V2.0,这个数据库注释的内容相对来说较少。同时。tRFTars在测序数据的使用上,使用了一个细胞系的CLASH数据以及一个CLIP数据。但是在靶基因预测上,在以上的数据基础上做作者使用了SVM这类的机器学习的算法来进行预测。
数据库使用
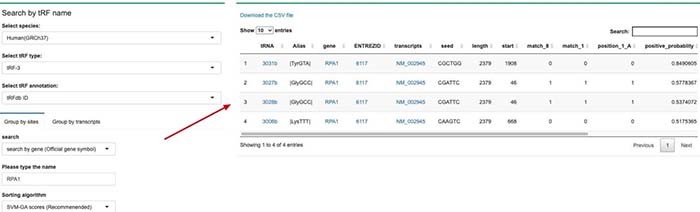
数据库的使用相对来说比之前的简单了。这个数据库主要就是用来预测靶基因的。所以我们在输入相对应的tRFs/基因之后。就可以获得相对应的结果了
总的来说
以上就是两个用来预测tRFs靶基因的数据库。由于使用的是不同的tRFs序列参考数据库也没办法比较说那个可能更好一些。但是如果是从分析的数据量来说的话,相对来说还是tRFTar更大一些。如果有想要研究tRF的童鞋可以尝试一下这两个数据库哈。














