由于序列较短和研究技术的限制,小蛋白在此前的基因组注释中通常被忽略。此外,虽然基因组中有丰富的sORF,但经过充分研究的小蛋白数量非常有限,其遗传学功能与角色仍存在着许多未知。因此,我们亟需对小蛋白进行系统的挖掘与注释,以促进对非编码RNA和基因组的全面了解,并为各种生理和病理过程的研究提供参考。
为此,中国科学院生物物理研究所陈润生院士团队和何顺民研究员团队合作在国际学术期刊Genomics, Proteomics & Bioinformatics在线发表了题为“SmProt: A Reliable Repository with Comprehensive Annotation of Small Proteins Identified from Ribosome Profiling”的文章(图1),介绍了该团队关于新版小蛋白数据库SmProt(http://bigdata.ibp.ac.cn/SmProt/)(图2)的工作,旨在提供关于小蛋白丰富、可靠的系统性注释。

图2. SmProt数据库。来源:Genomics, Proteomics & Bioinformatics
SmProt基于对419套公共核糖体图谱测序(ribosome profiling, Ribo-seq)数据的严格质控与重新分析,对已发表文献、数据库的信息挖掘,鉴定了来自人(Homo sapiens)、小鼠(Mus musculus)、大鼠(Rattus norvegicus)、果蝇(Drosophila melanogaster)、线虫 (Caenorhabditis elegans)、酵母(Saccharomyces cerevisiae)、斑马鱼(Danio rerio)、大肠杆菌(Escherichia coli)8个物种300余种组织/细胞系的3,165,229条小蛋白翻译事件记录,并收集了从人类微生物组中鉴定的小蛋白家族。通过对各种来源信息的交叉整合以及对结果的合并去冗余,获得了638,958个唯一的小蛋白,包括大量由UTR、非编码RNA编码的小蛋白(图3)。
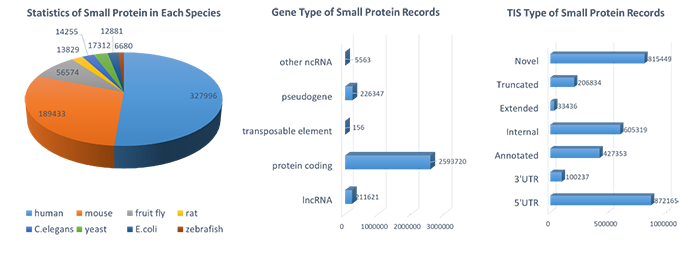
图3. SmProt中的小蛋白统计数据。来源:Genomics, Proteomics & Bioinformatics
SmProt主要基于肽酰基位点(peptidyl-tRNA site, P-site)偏移谱的三碱基周期性特征保证所鉴定小蛋白翻译事件的可靠性:
首先,研究团队基于独立发布的工具Ribo-TISH构建了新的翻译事件解析系统,允许使用常规Ribo-seq(regular Ribo-seq, rRibo-seq)和翻译起始测序(translation initiation sequencing, TI-seq)准确检测ORF和TIS。其使用秩和检验检测三碱基周期性,使用负二项分布检验检测翻译起始位点(translation initiation site, TIS),预测精度优于其他已建立的方法。
其次,除了基于Ribo-TISH quality模块的质量控制外,对所有数据集进行人工校验,以确保Ribo-seq数据具有清晰的三碱基周期性和明确的P-site偏移量,进一步消除噪声。
第三,提供多水平的支持证据,包括 (1)多个Ribo-seq数据集鉴定结果的P value,代表不同样本和处理条件下检测小蛋白的置信度;(2)相应基因组区域的PhyloCSF分值,反映其编码能力;(3)来自质谱数据的肽段证据。另外,基于文献数据库挖掘的小蛋白也与以上分析结果进行交叉整合,以互相验证。
同一物种中,从不同数据集和转录本鉴定出的相同基因组定位的sORF,被认为是相同的小蛋白,对其进行合并但保留不同数据集和转录本来源的信息。对于所鉴定的小蛋白,SmProt还进行了系统全面的注释,包括基本注释(图4)与功能性注释(图5):
1.SmProt整合多种来源的小蛋白信息,提供基本注释包括物种来源、组织/细胞系来源、数据来源、基因来源、长度、序列、基因组定位、起始密码子、编码基因区块、基因组区域编码能力、分子量,提供小蛋白的Ribo-seq分析信息、文献数据库收集信息、质谱检测肽段信息,并基于Ribo-seq数据提供其基因注释、翻译起始注释、显著性水平、翻译水平等信息。 2.基于蛋白序列分析,提供小蛋白功能域与蛋白家族注释,为功能性小蛋白的鉴定与研究提供参考依据。 3.基于翻译起始位点选择,提供小蛋白的不同亚型注释。 4.参考RNA-seq数据计算表达量RPKM (Reads Per Kilobase per Million mapped reads)的方式,基于Ribo-seq数据对每个sORF的阅读框内reads(in-frame reads)进行计数,并基于文库测序深度(样本in-frame reads总数)和sORF长度进行归一化,以代表sORF的翻译水平。 5.SmProt基于人类Ribo-seq数据鉴定了小蛋白编码序列上2万多个变异,及其对小蛋白的影响。有研究表明,上游ORF(upstream ORF, uORF)可通过泄漏扫描、重新启动和核糖体停滞等机制来调节下游 CDS的翻译。为促进对基因组调控机制的全面了解,SmProt集成多个全基因组测序(whole genome sequencing, WGS)项目资源,评估了人类全基因组变异对可能翻译的uORF及其下游蛋白编码序列(coding sequence, CDS)的影响。 6.进一步地,基于人类的Ribo-seq数据来源样本的表型信息,SmProt鉴定了包括肿瘤在内16种疾病中特异性存在的小蛋白及小蛋白上的变异,并基于已发表文献收录了已知的疾病相关小蛋白,为临床医学提供基于小蛋白视角的研究前景。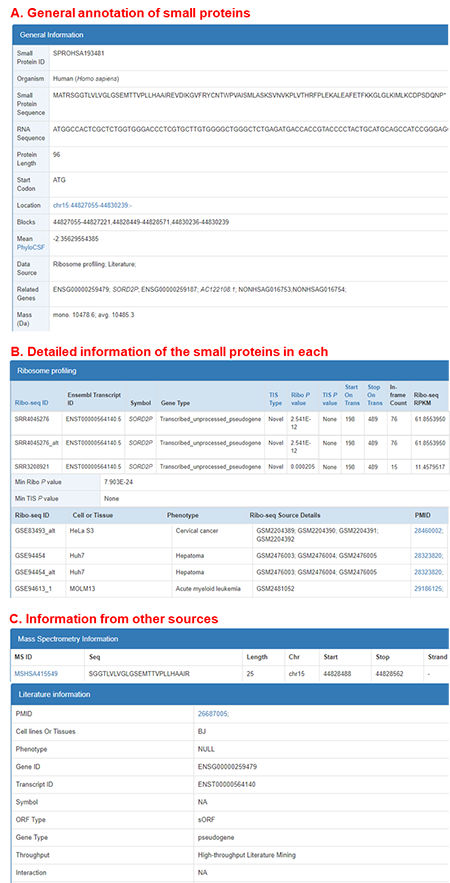
图4. 基于多种数据来源的小蛋白基本注释。 来源:Genomics, Proteomics & Bioinformatics
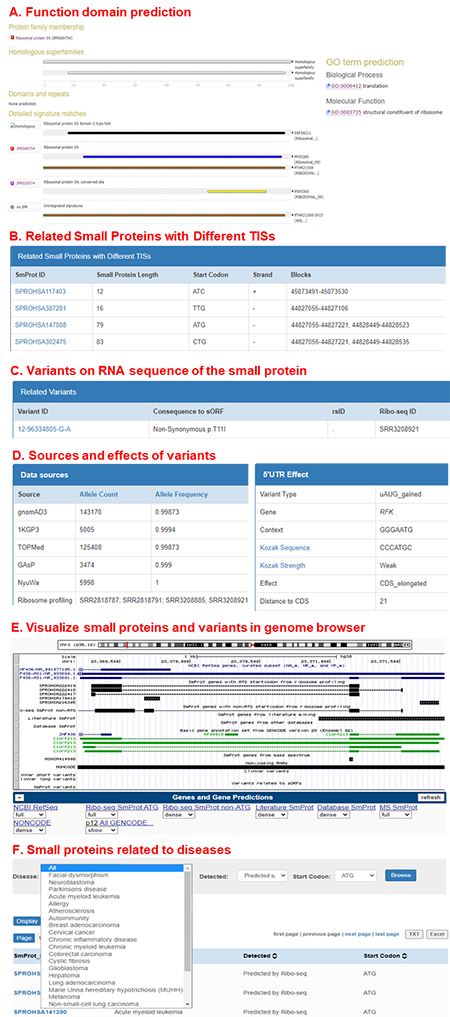
图5. 小蛋白的功能性注释。来源:Genomics, Proteomics & Bioinformatics
综上,SmProt对多个物种中小蛋白编码基因进行了系统性鉴定,进一步丰富了基因组注释,并通过全新的和多种来源、级别的翻译证据,确保结果的可靠性。当前SmProt版本提供了更加丰富全面的注释信息和功能模块,数据量、数据质量极大提升,并为非编码RNA研究、功能基因组学研究以及临床研究提供了新的参考。
中国科学院生物物理研究所的何顺民研究员、张鹏副研究员、陈润生院士为该文共同通讯作者,中国科学院大学的李燕燕、中国科学院生物物理研究所的周红红与陈晓敏为该文并列第一作者。该研究得到了国家重点研发计划、国家自然科学基金、中国科学院战略性先导科技专项(B类)、中国科学院信息化专项、科技部科技基础资源调查专项、国家基因组科学数据中心的支持。














