说到随机森林(random forest,RF),想必很多同学都不陌生了,毕竟这些机器学习方法目前非常流(fàn)行(làn)……白鱼同学也曾分别分享过“随机森林分类”以及“随机森林回归”在R语言中实现的例子,包括模型拟合、通过预测变量的值预测响应变量的值、以及评估哪些预测变量是“更重要的”等。在这两篇推文中,都是使用randomForest包执行的分析。不过在实际应用中,比方说想模仿一些文献的分析过程时,却发现某些统计无法通过randomForest包实现?
以评估预测变量的重要性为例,借助随机森林的实现方法经常在文献中见到,例如下面的截图所示。先前也有好多同学咨询,说如何像这篇文献中这样,计算出预测变量的显著性?虽说最常使用的randomForest包可以给出预测变量的相对重要性得分,允许我们根据得分排名从中确定哪些预测变量是“更重要的”,但却没有提供估计p值的方法。当我们出于某种需要想获知变量的显著性信息时,仅使用randomForest包就会很困扰?
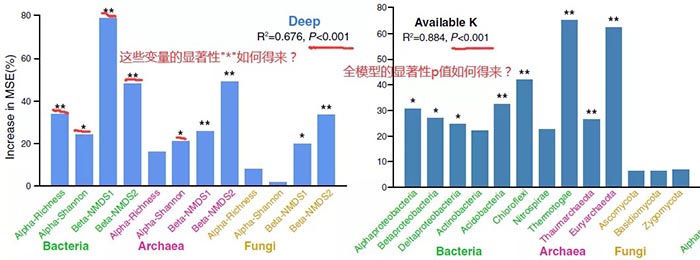
截图来自Jiao等(2018)的图5部分。左图展示了细菌、古细菌和真菌群落的α和β多样性在贡献深层土壤多养分循环指数中的重要性;右图展示了优势微生物分类群与土壤可利用钾的关系。两个图中变量的重要性以随机森林中的“percentage of increase of mean square error”(Increase in MSE(%))值进行衡量,更高的MSE%值意味着更重要的变量,并标识了各变量的显著性。图上方的数值为总方差解释率,以及全模型的显著性p值。
randomForest包实现不了的功能,那就用其它R包进行补充呗。至于用哪些R包可以,文献中通常都有详细的方法描述,仔细看一下材料方法部分大致就明确了。就以上面的Jiao等(2018)的文章为例,材料方法部分提到可通过A3包可获取对全模型显著性的估计,并可通过rfPermute包可获取对随机森林中预测变量重要性的显著水平估计。

接下来,就简单展示A3包和rfPermute包的使用,包括如何使用这些包执行随机森林分析,以及获取对全模型或者重要预测变量的显著性的估计。
下文的测试数据,R代码等的百度盘链接(提取码,z8zb):
https://pan.baidu.com/s/1-L78HuRzZCvH2LCzys4wJQ
若百度盘失效,也可在GitHub的备份中获取:
https://github.com/lyao222lll/sheng-xin-xiao-bai-yu
通过R包randomForest执行随机森林回归
为了进行对比说明,先来回顾一个先前的例子。
例如前文“随机森林回归”中使用R语言randomForest包执行随机森林回归。我们基于45个连续生长时间中植物根际土壤样本中细菌单元(OTU)的相对丰度数据,通过随机森林拟合了植物根际细菌OTU丰度与植物生长时期的响应关系(即,随机森林回归模型构建),根据植物根际细菌OTU丰度预测植物生长时期(即,通过预测变量对响应变量的值进行预测),并筛选出10个重要的具有明显时间特征的植物根际细菌OTU(即,评估预测变量的相对重要性并筛选重要的预测变量组合)。完整分析过程可参考前文“随机森林回归模型以及对重要变量的选择”,这里作了删减和改动,仅看其中的评估变量重要性的环节部分。
示例数据
网盘示例数据“otu_top10.txt”中,共记录了45个连续生长时间中植物根际土壤样本中10种细菌OTU的相对丰度信息。
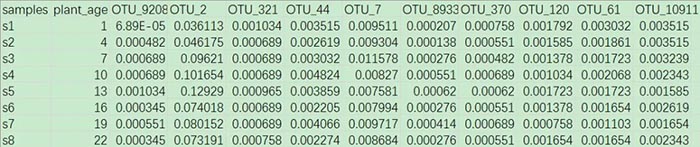
其中,samples列为45个样本的名称;plant_age记录了这45个根际土壤样本对应的植物生长时间(或称植物年龄),时间单位是天;其余10列为10种重要的细菌OTU的相对丰度信息,预先根据某些统计方法筛选出来的,它们已知与植物生长时间密切相关。
执行随机森林评估变量重要性
在这里,我们期望通过随机森林拟合这10种根际细菌OTU丰度与植物生长时期的响应关系,以得知哪些根际细菌OTU更能指示植物年龄。
#读取 OTU 丰度表
#包含预先选择好的 10 个重要的 OTU 相对丰度以及这 45 个根际土壤样本对应的植物生长时间(天)
otu <- read.delim('otu_top10.txt', row.names = 1)
##randomForest 包的随机森林
library(randomForest)
#随机森林计算(默认生成 500 棵树),详情 ?randomForest
set.seed(123)
otu_forest <- randomForest(plant_age~., data = otu, importance = TRUE, ntree = 500)
otu_forest
#使用函数 importance() 查看表示每个预测变量(细菌 OTU)重要性的得分(标准化后的得分)
importance_otu.scale <- data.frame(importance(otu_forest, scale = TRUE), check.names = FALSE)
importance_otu.scale
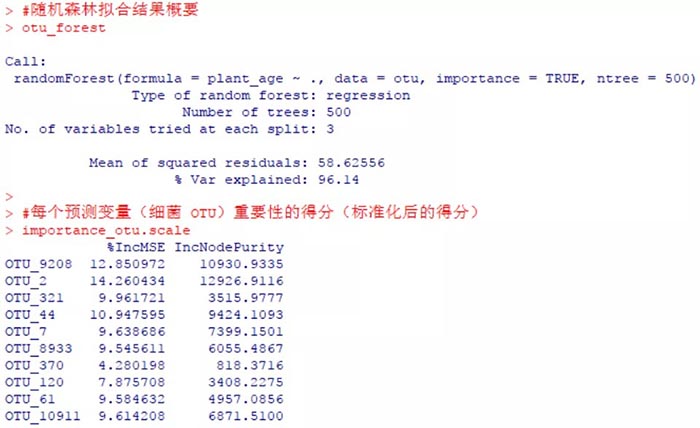
如前所述,结果中“% Var explained”体现了预测变量(用于回归的10个细菌OTU)对响应变量(植物年龄)有关方差的整体解释率,这里为96.14%,反映了这个随机森林模型很高的拟合优度。
函数importance()给出了预测变量(10个细菌OTU)的相对重要性得分。“%IncMSE”即increase in mean squared error,通过对每一个预测变量随机赋值,如果该预测变量更为重要,那么其值被随机替换后模型预测的误差会增大。“IncNodePurity”即increase in node purity,通过残差平方和来度量,代表了每个变量对分类树每个节点上观测值的异质性的影响,从而比较变量的重要性。两个指示值均是判断预测变量重要性的指标,均是值越大表示该变量的重要性越大,但分别基于两者的重要性排名存在一定的差异。至于选择哪一个更合适,自己看着来吧。
最后绘制柱形图观察和比较这些预测变量(10个细菌OTU)的相对重要性得分及排名。
#对预测变量(细菌 OTU)按重要性得分排个序,例如根据“%IncMSE”
importance_otu.scale <- importance_otu.scale[order(importance_otu.scale$'%IncMSE', decreasing = TRUE), ]
#简单地作图展示预测变量(细菌 OTU)的 %IncMSE 值
library(ggplot2)
importance_otu.scale$OTU_name <- rownames(importance_otu.scale)
importance_otu.scale$OTU_name <- factor(importance_otu.scale$OTU_name, levels = importance_otu.scale$OTU_name)
p <- ggplot(importance_otu.scale, aes(OTU_name, `%IncMSE`)) +
geom_col(width = 0.5, fill = '#FFC068', color = NA) +
labs(title = NULL, x = NULL, y = 'Increase in MSE (%)', fill = NULL) +
theme(panel.grid = element_blank(), panel.background = element_blank(), axis.line = element_line(colour = 'black')) +
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
scale_y_continuous(expand = c(0, 0), limit = c(0, 16))
p
#右上角备注模型的已知解释率
p <- p +
annotate('text', label = 'Plant Age', x = 9, y = 15, size = 4) +
annotate('text', label = sprintf('italic(R^2) == %.2f', 96.14), x = 9, y = 13, size = 3, parse = TRUE)
p
通过rfPermute包执行随机森林回归以及获取变量的显著性
尽管上文randomForest包通过计算预测变量的相对重要性得分,允许我们根据得分排名从中确定预测变量的可靠程度,但没有告知我们这些变量是否是显著的。毕竟有些情况下我们确实想迫切知道变量的显著性,如Jiao等(2018)里的那样(本文开篇截图所示文献),因为这些统计量在这些情况中可能很有用。但由于randomForest()函数没有提供估计p值的方法(虽然它有个参数nPerm,但很可惜并不是计算p值的功能),就会很困扰。
仿照Jiao等(2018)的方法,我们可以使用rfPermute包的随机森林去评估每个预测变量(用于回归的10个细菌OTU)对响应变量(植物年龄)的重要性,并获得显著性信息。其实在使用过程中不难看出,rfPermute包沿用了randomForest包的随机森林方法,并对randomForest包的功能作了一些拓展。事实上,我们其实可以跳过randomForest包,直接通过rfPermute包对上文给定的数据执行随机森林分析,会得到和randomForest包一样的运行结果。然后rfPermute包的优势在于给出预测变量重要性得分的同时,还基于置换检验的原理对重要性得分进行了检验,并提供了显著性信息。
library(rfPermute)
#使用函数 rfPermut() 重新对上述数据执行随机森林分析,详情 ?rfPermut
#rfPermut() 封装了 randomForest() 的方法,因此在给定数据和运行参数一致的情况下,两个函数结果也是一致的
#并在这里额外通过 nrep 参数执行 1000 次的随机置换以评估变量显著性的 p 值
#若数据量较大,可通过 num.cores 参数设置多线程运算
set.seed(123)
otu_rfP <- rfPermute(plant_age~., data = otu, importance = TRUE, ntree = 500, nrep = 1000, num.cores = 1)
otu_rfP
#提取预测变量(细菌 OTU)的重要性得分(标准化后的得分)
importance_otu.scale <- data.frame(importance(otu_rfP, scale = TRUE), check.names = FALSE)
importance_otu.scale
#提取预测变量(细菌 OTU)的重要性得分的显著性(以标准化后的得分为例)
# summary(otu_rfP)
importance_otu.scale.pval <- (otu_rfP$pval)[ , , 2]
importance_otu.scale.pval
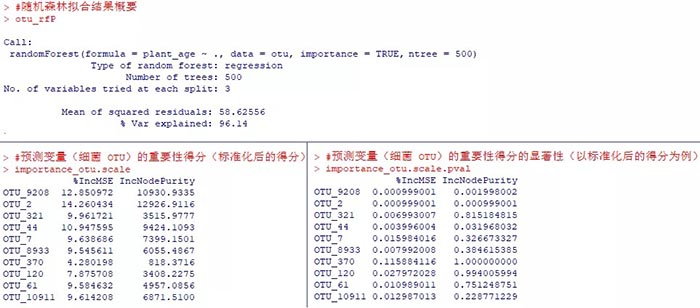
我们看到rfPermute()的结果中“% Var explained”为96.14%,和上文randomForest()的结果完全一致。但rfPermute()除了给出了预测变量(10个细菌OTU)的相对重要性得分“%IncMSE”和“IncNodePurity”外,还估计了得分的显著性信息,这是randomForest()所没有提供的。
类似地,基于两个指示值的重要性排名和显著性存在一定的差异,实际中二选一看着来。
#作图展示预测变量(细菌 OTU)的重要性得分(标准化后的得分),其中显著的得分(默认 p<0.05)以红色显示
plot(rp.importance(otu_rfP, scale = TRUE))
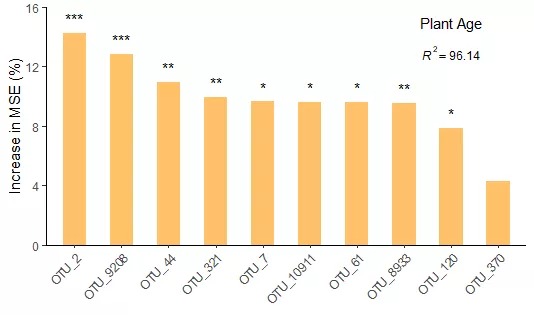
这次,我们即可将这些预测变量(10个细菌OTU)的显著性信息添加在上文的柱形图中,和它们的相对重要性得分及排名一起展示。
#对预测变量(细菌 OTU)按重要性得分排个序,例如根据“%IncMSE”
importance_otu.scale <- importance_otu.scale[order(importance_otu.scale$'%IncMSE', decreasing = TRUE), ]
#简单地作图展示预测变量(细菌 OTU)的 %IncMSE 值
library(ggplot2)
importance_otu.scale$OTU_name <- rownames(importance_otu.scale)
importance_otu.scale$OTU_name <- factor(importance_otu.scale$OTU_name, levels = importance_otu.scale$OTU_name)
p <- ggplot() +
geom_col(data = importance_otu.scale, aes(x = OTU_name, y = `%IncMSE`), width = 0.5, fill = '#FFC068', color = NA) +
labs(title = NULL, x = NULL, y = 'Increase in MSE (%)', fill = NULL) +
theme(panel.grid = element_blank(), panel.background = element_blank(), axis.line = element_line(colour = 'black')) +
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
scale_y_continuous(expand = c(0, 0), limit = c(0, 16))
p
#标记预测变量(细菌 OTU)的显著性信息
#默认以 p<0.05 为 *,p<0.01 为 **,p<0.001 为 ***
for (OTU in rownames(importance_otu.scale)) {
importance_otu.scale[OTU,'%IncMSE.pval'] <- importance_otu.scale.pval[OTU,'%IncMSE']
if (importance_otu.scale[OTU,'%IncMSE.pval'] >= 0.05) importance_otu.scale[OTU,'%IncMSE.sig'] <- ''
else if (importance_otu.scale[OTU,'%IncMSE.pval'] >= 0.01 & importance_otu.scale[OTU,'%IncMSE.pval'] < 0.05) importance_otu.scale[OTU,'%IncMSE.sig'] <- '*'
else if (importance_otu.scale[OTU,'%IncMSE.pval'] >= 0.001 & importance_otu.scale[OTU,'%IncMSE.pval'] < 0.01) importance_otu.scale[OTU,'%IncMSE.sig'] <- '**'
else if (importance_otu.scale[OTU,'%IncMSE.pval'] < 0.001) importance_otu.scale[OTU,'%IncMSE.sig'] <- '***'
}
p <- p +
geom_text(data = importance_otu.scale, aes(x = OTU_name, y = `%IncMSE`, label = `%IncMSE.sig`), nudge_y = 1)
p
#右上角备注模型的已知解释率
p <- p +
annotate('text', label = 'Plant Age', x = 9, y = 15, size = 4) +
annotate('text', label = sprintf('italic(R^2) == %.2f', 96.14), x = 9, y = 13, size = 3, parse = TRUE)
p
通过A3包获取全模型的显著性
另一方面,randomForest包虽然给出了全模型的方差解释率(即,R2),但也没有对全模型的显著性进行评估。不过与上述各个预测变量的p值相比,全模型的p值倒不是很纠结人,因为根据经验,只要R2不是特别小,p值都是绝对显著的。例如这里R2=0.9614,用眼睛就能直接判断出来p<0.001……当然话虽这样说,该计算的还是要计算一下。
同样仿照Jiao等(2018)的方法,我们可以使用A3包评估全模型的显著性。
library(A3)
#model.fn=randomForest 调用随机森林的方法进行运算
#p.acc=0.001 表示基于 1000 次随机置换获得对 p 值的估计,p.acc 值越小代表置换次数越多,运算也就越慢,因此如果对全模型 p 值不是很迫切的话还是慎用
#model.args 用于传递参数给 randomForest(),因此里面的参数项根据 randomForest() 的参数项而定,具体可 ?randomForest
#其它详情可 ?a3 查看帮助
set.seed(123)
otu_forest.pval <- a3(plant_age~., data = otu, model.fn = randomForest, p.acc = 0.001, model.args = list(importance = TRUE, ntree = 500))
otu_forest.pval

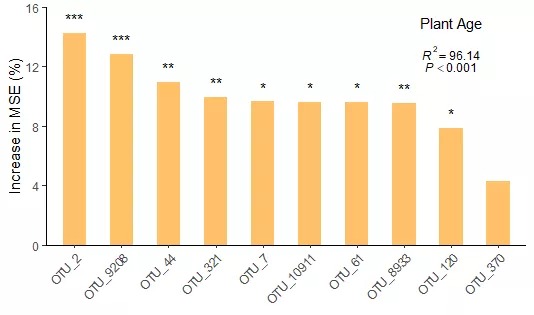
我们看到全模型的p<0.001是非常显著的。由于随机的因素在里面,这里的R2和上文的R2相比有很微小的差异,但是并无大碍,就默认为它们一致就可以了。至于结果中的其它值反映了什么信息,我没有过多关注,大家有兴趣可以自己研究下。
其它缺点是这一步非常耗时,貌似还不能多线程,因此大数据慎用……
最后,我们再将全模型的显著性信息备注在上文的柱形图中,和全模型的方差解释率、预测变量(10个细菌OTU)的相对重要性得分、排名以及显著性信息一起展示。
#继续在右上角备注全模型的显著性信息
p <- p +
annotate('text', label = sprintf('italic(P) < %.3f', 0.001), x = 9, y = 12, size = 3, parse = TRUE)
p
参考资料
Jiao S, Chen W, Wang J, et al. Soil microbiomes with distinct assemblies through vertical soil profiles drive the cycling of multiple nutrients in reforested ecosystems. Microbiome, 2018, 6(1): 1-13.
rfPermute包:https://www.rdocumentation.org/packages/rfPermute/versions/2.1.81
A3包:https://rdrr.io/cran/A3/
文章来源于小白鱼的生统笔记














