今天给大家解读一篇2022年3月发表在E BioMedicine(IF:8.143)上的文章。本研究构建了一个计算框架可以鉴定药物作用的潜在靶点并构建一个药物重定位方法鉴定潜在靶点的有效药物。
Prediction of drug candidates for clear cell renal cell carcinoma using a systems biology-based drug repositioning approach
基于系统生物学的药物重定位方法预测肾透明细胞癌的候选药物
摘要
背景:肾透明细胞癌(ccRCC)患者对临床化疗的效果较低,药物重定位方法作为一种有前途的策略可以有效的鉴定候选药物,对无法获益于临床化疗的患者具有一定作用。
方法:作者提出了一种基于ccRCC患者转录组数据的共表达网络分析和肾细胞系shRNA-和药物干扰特征谱分析的癌症治疗药物重定位方法。
结果:基于基因共表达网络分析,鉴定到两个基因模块,分别与患者生存较差和较好有关。随后,根据模块的拓扑分析鉴定到4个潜在药物靶点,分别为BUB1B, RRM2, ASF1B和CCNB2。通过药物重定位方法将三种最有效的药物应用于4个靶点。使用体外模型评估药物的作用,结果表明这些药物抑制了相应靶基因的蛋白水平和细胞活力。
流程图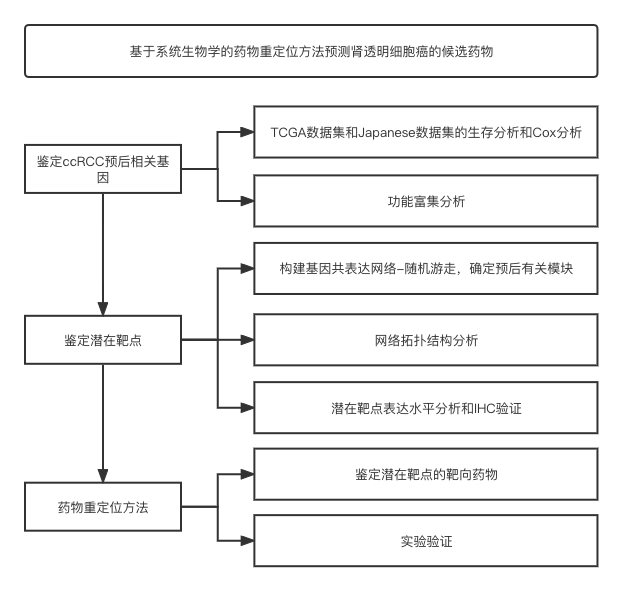
结果
1. 数据集的获取和下载
从https://osf.io/gqrz9 获取TCGA数据集包括528例ccRCC样本和72例正常样本的RNA-seq数据。从European Genome-phenome Archive获取100例ccRCC样本的RNA-seq数据。从DepMap Portal获取16个ccRCC细胞系的基因打分数据。
2. 鉴定ccRCC特征基因
对TCGA数据集和Japanese数据集进行生存分析和单因素Cox回归分析,TCGA数据集鉴定到7813个预后基因,而Japanese数据集鉴定到1335个预后基因,两个基因集取交集共有1129个基因(图1a)。这1129个基因中有787个基因与预后较好有关而有342个基因与预后较差有关。功能富集分析表明,与预后较差有关的基因显著富集于细胞分裂和细胞循环通路,而与预后较好有关的基因显著富集于血管生成,细胞迁移和细胞分化等通路(图1b)。
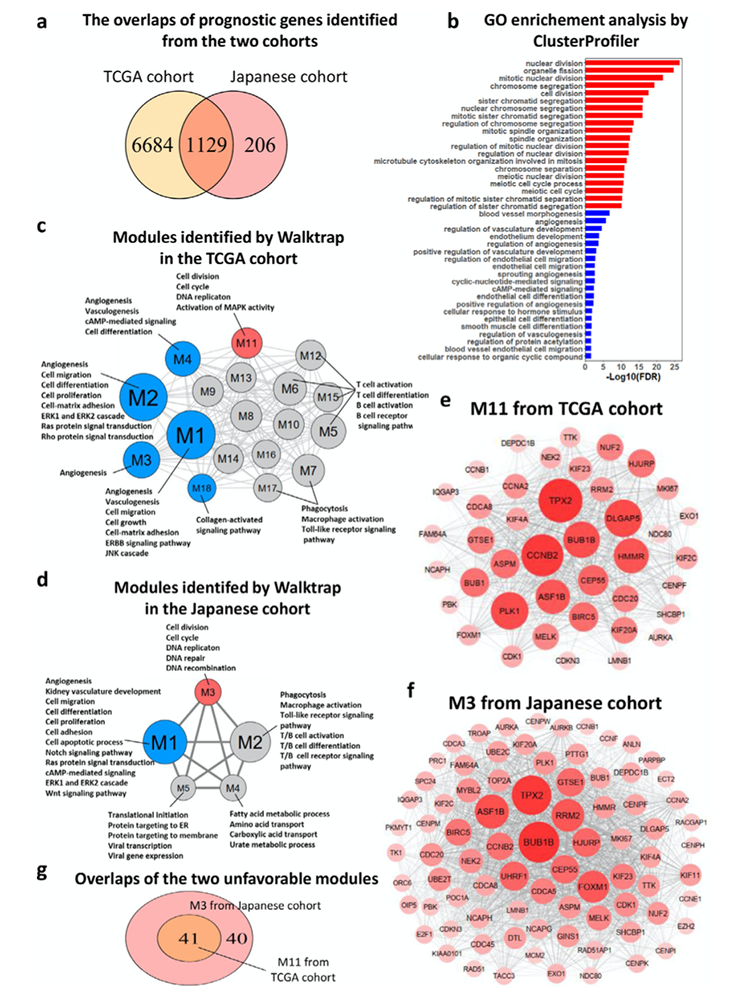
图1 鉴定ccRCC患者的特征基因和功能基因模块
3. GCN分析鉴定功能模块
GCN方法可以有效鉴定驱动关键细胞信号通路的基因,包括参与肿瘤发生过程的基因。首先,作者计算TCGA数据集和Japanese数据集各个基因对的Spearman相关系数,并保留相关性排名前1%的基因对,即构建GCN。随后,使用随机游走算法鉴定关键基因模块。为了鉴定具有功能意义的基因模块,作者仅保留模块中基因数量大于20个的模块且聚类相关性大于0.6。最终,TCGA数据集获得18个模块(M1-18)(图1c)。将预后相关基因与基因模块取交集,其中M11模块中有40个与预后较差的基因,因此将M11模块定义为与预后较差有关的模块。M1,M2,M3,M4和M18模块中的基因与预后较好有关的基因重复较多,因此将M1,M2,M3,M4和M18模块定义为预后较好有关的模块。富集分析表明,M11模块中的基因显著富集于细胞分裂,细胞循环,DNA富集和MAPK活化通路(图1c)。M1,M2,M3,M4和M18模块中的基因显著富集于血管生成,细胞迁移,细胞分化和cAMP介导信号通路等(图2c)。对Japanese数据集进行同样分析,得到5个模块(M1-5)(图1d)。其中,M1定义为预后较差有关的模块而M3定义为预后较好有关的模块(图1d)。基于以上结果,TCGA数据集中的模块M11和Japanese数据集中的模块M3是预后较差有关的模块,而TCGA数据集中的模块M1,M2,M3,M4,M18和Japanese数据集中的模块M1是预后较差有关的模块是预后较好有关的模块。图1e和1f分别为TCGA数据集中的模块M11和Japanese数据集中的模块M3的共表达网络。TCGA数据集中的模块M11和Japanese数据集中的模块M3中的基因重叠较多(图1g)。以上结果表明,作者基于两个独立数据集鉴定到了与预后有关的基因模块。
4. 鉴定潜在的药物作用靶点基因
网络拓扑结构分析可以研究网络中基因的重要性,中心度较好的基因可能是网络中的核心基因,这些基因可以作为潜在靶点。作者分别计算TCGA数据集和Japanese数据集中与预后有关的模块中基因的中心性和Spearman相关性(图2a和2b)。TCGA数据集和Japanese数据集中与预后较差有关模块的度中心性,中介中心性和接近中心性的相关性分别为0.77,0.73和0.78(图2a)。TCGA数据集和Japanese数据集中与预后较好有关模块的度中心性,中介中心性和接近中心性的相关性分别为0.73,0.71和0.72(图2b)。以上结果表明,TCGA数据集和Japanese数据集鉴定到的模块的拓扑结构相似性较高。随后,根据度中心性,中介中心性和接近中心性对各个模块中的基因进行排序,将预后较差有关模块中排名前10位的基因取交集,获得6个核心基因分别为BUB1B, TPX2, RRM2, CEP55, ASF1B和CCNB2(图2c)。将预后较好有关模块中排名前10位的基因取交集,获得4个核心基因分别为ERG, ARAP3, TCF4和MMRN2(图2d)。
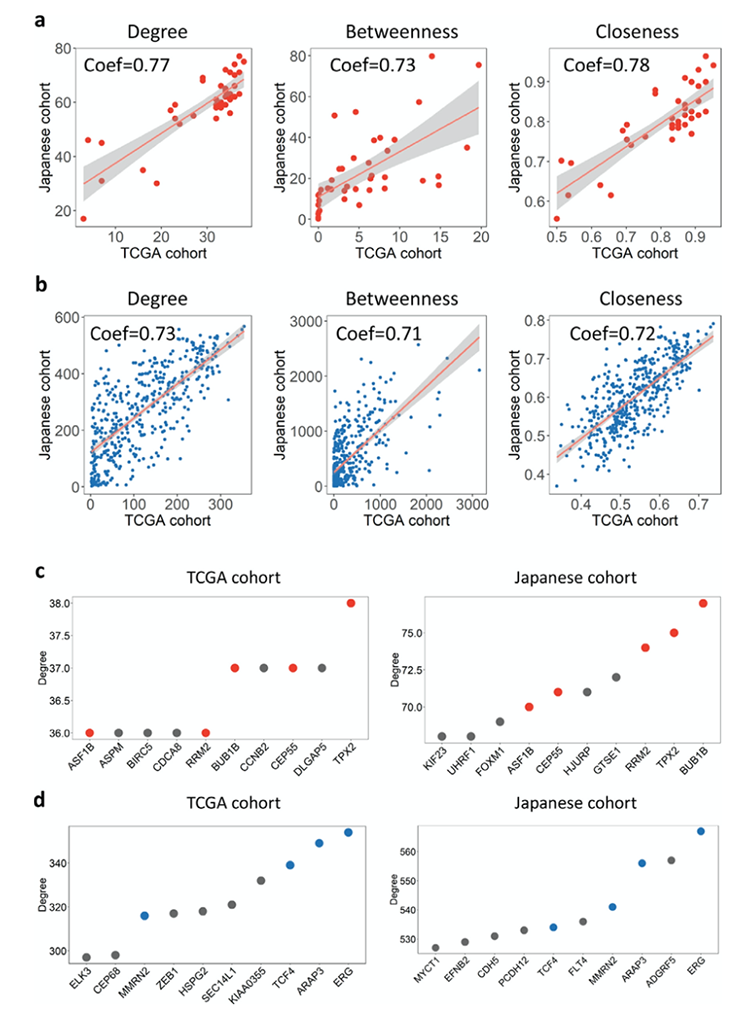
图2 模块的拓扑结构分析
随后,作者分析TCGA数据集中癌症样本和正常样本中这10个基因的表达水平,大多数基因在正常组织中低表达而在癌症组织中高表达(图3a和3b)。作者进一步研究各个肿瘤组织中核心基因的表达情况,有84-86%的样本发生预后较差基因高表达且生存期较差,有59-73%的样本发生预后较好基因高表达且生存期较好(图3c)。此外,使用IHC验证基因的表达水平,其中有4个基因在肿瘤组织中蛋白表达水平较好(图3d)。从16个ccRCC细胞细胞系的基础打分数据集中提取这些核心基因的基础打分。一个基因的打分越低表明这个基因对肿瘤细胞增殖和生存越重要。如图3e所示,大部分预后较差的核心基因的打分较低。作者同样研究这些核心基因在Caki-1中的表达水平,预后较差的核心基因表达水平较高(图3f)。
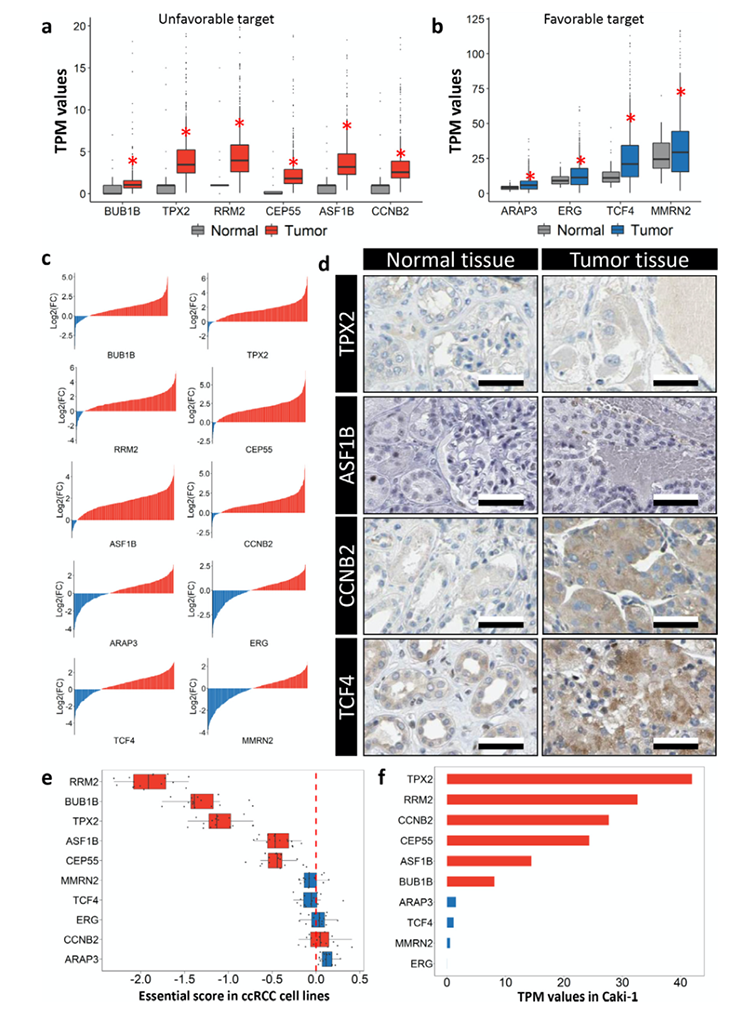
图3 核心基因的表达水平
5. 靶基因的药物重定位
为鉴定候选药物靶点的潜在治疗药物,作者构建了一种基于药物和shRNA扰动特征图谱的综合分析的药物重定位方法。作者认为如果药物治疗导致了类似由shRNA敲除引起的基因表达失调,则药物对肿瘤细胞中靶基因的表达具有抑制作用。作者构建的药物重定位方法如图4a所示,主要包括以下四个步骤:首先,构建靶基因的药物-shRNA矩阵;其次,通过设置剂量和处理时间优化药物-shRNA矩阵;第三,提取排名前1%的候选药物;最后,对每个基因靶点选择最有效的三种药物(图4a)。基于上述方法,作者鉴定到TG-101209, oxetane,和WH-4-025靶向BUB1B, NVP-TAE684和MK-0752,withaferin-a靶向RRM2,actinomycin-d, triphenyl-tin,和RS-I-002-6靶向CEP55,和BRD-K26510616,panobinostat和tacedinaline靶向ASF1B,oxetane和BRD-K54343811 靶向CCNB2(图4b)。
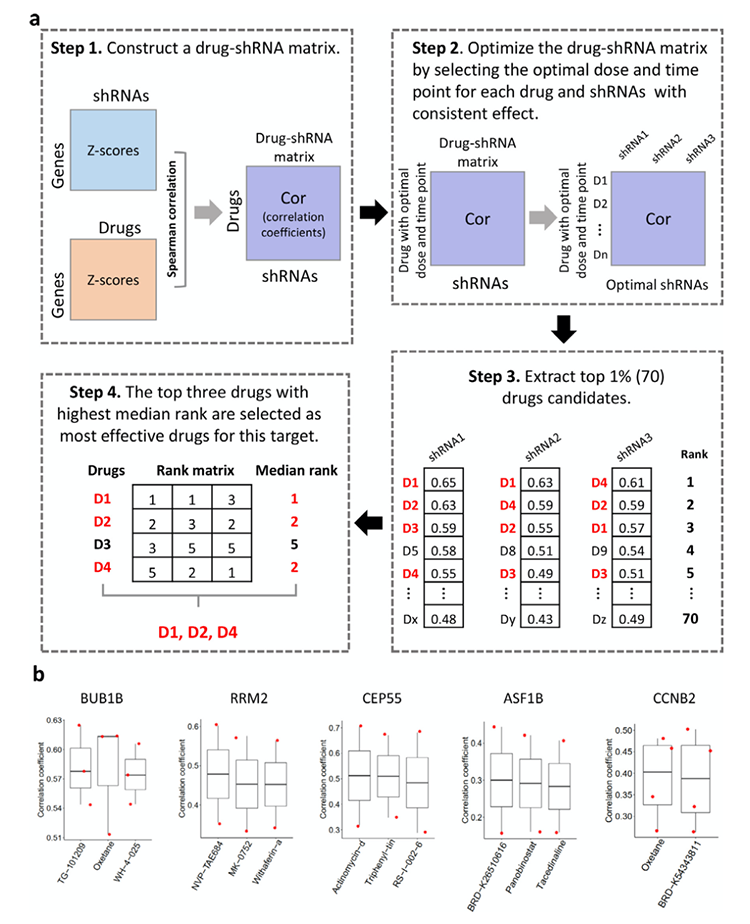
图4 药物重定位方法
6. 验证靶基因的重要性和药物影响
为验证靶基因在ccRCC中的重要性,作者通过siRNAs转染Caki-1抑制靶基因的表达并研究对细胞活力的影响。Western blot结果表明,siRNAs转染Caki-1成功抑制了5个靶基因的蛋白表达(图5a-5e)。使用CCK-8实验测量细胞增殖情况,结果表明Caki-1中敲除BUB1B、RRM2、ASF1B和CCNB2会显著降低细胞活力,而敲除CEP55不会显著降低细胞活力(图5a-5e)。此外,作者研究药物是否会抑制相应靶基因。结果表明,TG-101209处理组BUB1B蛋白水平显著降低且细胞活力显著降低,因此TG-101209是靶向BUB1B最有效的药物(图5f)。NVP-TAE684、、和withaferin-a处理会显著抑制RRM2的蛋白表达水平和细胞活力,而MK-0752并没有抑制RRM2的蛋白表达水平(图5g)。此外,作者研究actinomycin-d对RRM2的作用,Western blot结果表明actinomycin-d不会抑制RRM2的表达。panobinostat靶向ASF1B会显著抑制ASF1B的蛋白表达水平且细胞活性显著降低(图5h)。总的来说,作者构建的药物重定位方法可以鉴定有效的候选药物并确定了BUB1B、RRM2和ASF1B的药物靶点。
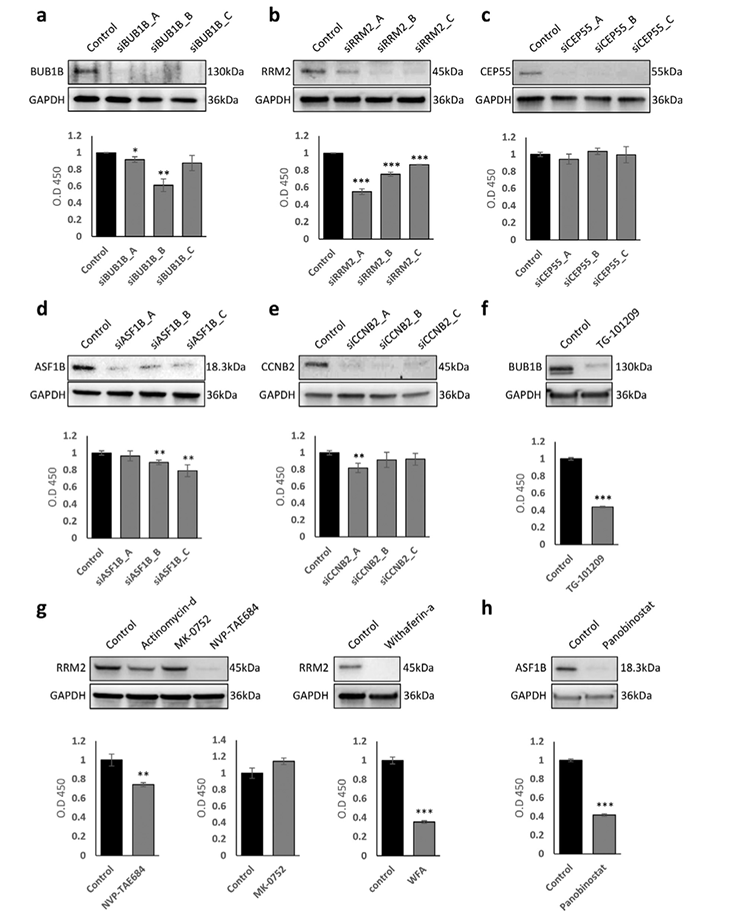
图5 验证靶基因和药物作用
结论
总的来说,本研究对两个转录组数据集进行分析构建基因共表达网络并基于随机游走算法鉴定与预后有关的模块,根据网络拓扑结构鉴定潜在靶点。随后,作者构建药物重定位计算框架并鉴定潜在靶点的靶向药物。最后,对潜在靶点和药物作用进行实验验证。本研究的亮点在于作者的分析方法,作者构建了两个计算框架可以用于ccRCC患者鉴定潜在靶点和靶向药物。本研究的局限性在于这两个计算框架是否可以应用于其他癌症类型还需要进一步研究。













