
GWAS位点90%以上分布在非编码区域,目前这些位点潜在发挥功能的机制依然很不清楚(1)。
为此,纽约基因组中心(New York Genome Center)Neville Sanjana以及Tuuli Lappalainen等研究人员基于CRISPR筛选和单细胞多组学开发-STING-seq(Systematic Targeted Inhibition of Noncoding GWAS loci coupled with single-cell sequencing),并研究了部分经前期分析高度与表型有因果关联的非编码GWAS位点(更准确来讲是这些关联位点所在的区域)靶向的基因与生物学通路(1, 2)。
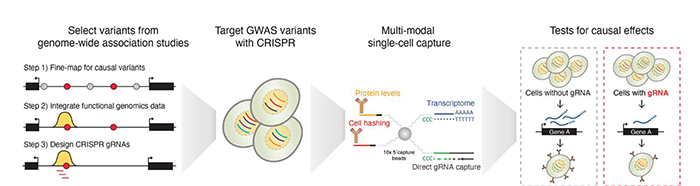 基于单细胞CRISPR筛选的STING-seq平台分析非编码GWAS位点的靶向基因(1)
基于单细胞CRISPR筛选的STING-seq平台分析非编码GWAS位点的靶向基因(1)
研究人员通过该平台发现了91个与血液表型相关的非编码区域直接(或者说顺式,cis-regulation)靶向的124个基因;并进一步分析了部分非编码区域通过调节邻近转录因子或miRNA表达引发的反式调控网络(trans-regulatory networks)变化(也就是非编码区域变异-->顺式调节邻近的转录因子或miRNA表达-->转录因子/miRNA通过靶向序列结合调节更大范围的基因表达)(1)。
该项工作2023年5月4日发表在Science;研究人员表示该研究为研究非编码区域功能机制打下基础,并有望带来新疗法。
Comment(s):
该项工作提出的STING-seq平台扩展性比较高,可以快速应用于其他类似科学问题。
但是,将来还是要基于更精准的base editor/prime editing技术(当然首先需要进一步提高其效率)直接在与科学问题关联更强的细胞系或者原代细胞研究GWAS变异的功能影响(3, 4); 或者,另一个方向,做到全基因组水平的非编码序列靶向基因/通路解析。













