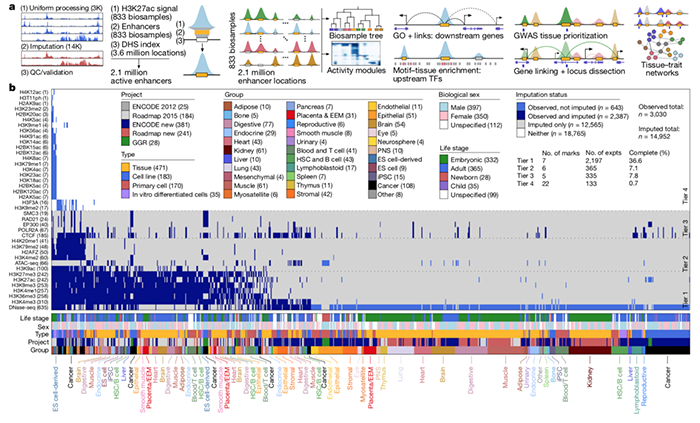
近日,来自MIT和Broad中心的Manolis Kellis 课题组在Nature上发表了文章Regulatory genomic circuitry of human disease loci by integrative epigenomics,绘制了迄今最全面的与疾病相关的表观遗传图谱,并命名为EpimMap (Epigenome Integration across Multiple Annotation Projects)。尽管此前已经有了Encode【2】, Roadmap【3】等基因组注释数据库, 但他们仍有不足之处:缺少疾病相关联组织的数据,因实验室、方法、抗体批次等差异造成的数据质量参差不齐、生物样品及表观遗传标记覆盖度差异(仅在少数生物样品中的多种表观遗传标记或大量生物样品却只有少数标记)。这篇文章目标就是克服这些问题, 具体来说:整合了Encode、Roadmap、以及Genomics of Gene Regulation (GGR)【4】 注释图谱数据库的833个生物样品(剔除掉低质量数据),涵盖了包括H3K4me1, H3K4me3, H3K27ac, H3K36me3, H3K9me3,H3K27me3 多种组蛋白修饰、Rad21、CTCF等Chip-seq数据,体现DNA开放性的Dnase-seq, ATAC-seq等18种表观遗传标记,绘制了表观遗传图谱(图1)。作者进一步利用这些高分辨率的表观修饰数据定义染色体开放状态,高分辨率地注释了210,000个增强子,依据增强子的活性状态进行模块聚类,共产生300个增强子聚类模块,其中290个是组织特异性的,另外10个广谱活性的,实现增强子模块与组织关联。再依据增强子的motif序列推测上游调控因子(转录因子等),如在脑组织中NeuroD2和RFX4。利用机器学习预测增强子的下游靶基因,并对这些基因进行功能富集分析(Gene ontology)。接下来他们通过整合高分辨率的基因调控环路和基因关联结果,揭示了与性状相关的表观遗传富集。由此实现表观遗传标记、特异组织、性状、GWAS数据的整合分析。文章的最后还将此关联数据应用于分析多因素性状,如他们发现与冠心病(CAD)相关的表观遗传修饰的改变在脂肪组织,冠状动脉和肝脏以及许多其他组织中均有富集。文章的最后,作者也指出该工作的不足之处:生物样品不是单细胞分辨率;没有考虑供体的基因型或表型;推演计算可能增强了共性部分而漏掉了特异性,他们仍漏掉了很多组织、环境与刺激因素以及发育阶段。同时也指出了该工作的意义:“我们的工作将助力于未来的研究:基于层级和多层分辨率树的基因调控和GWAS分析;基于机器学习的基因环路和组合调控组块分析;组织-性状,性状-性状和组织-组织关系更复杂的网络分析;并指导实验的优先次序,方法学的发展和验证实验,这些都将推动我们对基因调控网络、疾病调控环路的研究”。这些数据都已公开在http://compbio.mit.edu/epimap https://www.nature.com/articles/s41586-020-03145-z