当前,仅有部分癌症患者能从化疗、靶向治疗和免疫治疗中获益。比如,一个大规模二期单药临床试验的荟萃分析(包含570个临床试验和32149个病人)表明化疗的中位响应率仅为11.9%,而个性化的靶向治疗的响应率仅为30%【1】。药物抵抗仍旧是现在癌症研究面临的主要且挑战性的问题。精准医学时代正快速积累大量具有临床用药响应信息的病人来源的临床转录组数据,这些数据为理解癌症病人用药抵抗背后的分子机制,确定病人治疗精准分层的标志物,甚至是发展能够克服抵抗的联合用药提供了宝贵资源。然而这些数据分散,且经常使用不同的术语、不一致的数据处理流程、有计算机可读性差的元信息等,为数据的综合利用带来了困难。
近日,国家蛋白质科学中心(北京)李栋研究员/贺福初院士团队在Nucleic Acids Research期刊发表论文“CTR-DB, an omnibus for patient-derived gene expression signatures correlated with cancer drug response”,报道了他们建立的首个具有临床用药响应信息的病人来源的癌症临床转录组数据资源CTR-DB(http://ctrdb.ncpsb.org.cn)。该资源为基础和临床研究者提供了一个接入、整合和重利用癌症治疗响应临床转录组数据的接口,助力癌症药物抵抗机制揭示、联合药物发现以及抵抗机制异质性探索,尤其是为利用病人转录组数据发现和验证药物响应预测标志物提供了重要资源(图1)。

图1 CTR-DB (来源:https://doi.org/10.1093/nar/gkab860)
数据方面,遵照严格的纳入标准,目前CTR-DB手工收集了来自于GEO,ArrayExpress和TCGA的83个具有用药响应信息的病人来源的基线转录组源数据集。为了方便数据整合和重利用,所有转录组数据集均采用统一的数据处理流程进行重处理(RNA-seq数据以FASTQ文件为起点,Microarray数据以CEL文件为起点);药物响应等样本元信息(包括癌症亚型、药物、药物响应状态及其注释、数据产出平台等)均通过手工挖掘获得,并进行了术语标准化。这些源数据集被进一步分成626个“CTR-DB数据集”,每个CTR-DB数据集由使用相同治疗方案且具有相同癌症亚型(最细颗粒度)的病人样本所组成的。共涉及275个治疗方案、123个药物、28个癌症组织类型和5139个样本(图2)。所有统一处理后的表达谱数据及其用药响应等元信息支持批量下载。
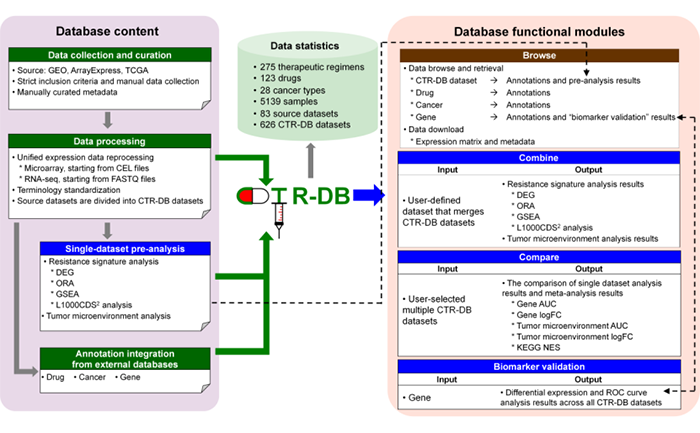
图2 CTR-DB数据库内容和功能模块概览
功能方面,为了方便数据整合和(重)利用,除了基本的浏览、检索和下载,CTR-DB支持多种数据分析功能/工作流(图1),且所有分析结果支持下载。文章以CTR-DB中三个与PD1/PD-L1治疗抵抗相关数据集作为例子,展示以下所有功能的使用和价值。
1) 单数据集分析
a) 非响应/响应组差异基因表达分析(图3A)、基因ROC曲线分析(图3A)、功能富集分析(图3B,C)工作流,旨在帮助理解药物抵抗机制,发现候选的药物响应预测标志物;
b) 搜索能够逆转药物A的抵抗信号(图3D)或者能够抑制药物A抵抗相关差异高表达基因(图3E)的药物B,旨在发现能够克服药物A抵抗的候选联合药物B;
c) 肿瘤微环境分析(图3F),旨在探索与药物抵抗相关的肿瘤微环境因素。
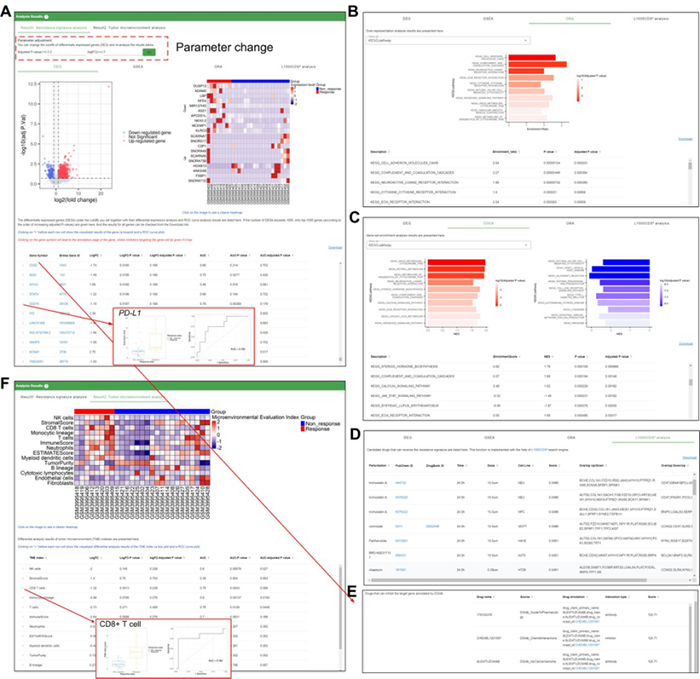
图3 单数据集分析结果页面
2) 多数据集联合分析
用户可以选择和联合多个CTR-DB数据集并根据需要自己指定响应/非响应分组。通过此功能,典型地,用户可以联合一类药物(比如PD1/PD-L1抑制剂)或一个更高颗粒度的癌症亚型的数据集。被支持的数据分析功能同上述单一数据集。
3) 多数据集比较
实现用户选择的多个CTR-DB数据集的分析结果的比较以及它们的荟萃分析(图4A),旨在探索不同数据集抵抗机制的异同,甚至发现可能的“泛数据集”共享的抵抗机制和预测标志物。
4) 药物响应预测标志物验证
药物响应预测标志物能够预测病人对药物的响应,对癌症精准治疗至关重要。对于某个药物治疗某个癌种的候选标志物,用户可以使用相应的CTR-DB病人群体的临床转录组数据验证其基因表达水平区分响应/不响应病人的能力;或者对于感兴趣的基因,用户可以查看其基因表达与各个药物抵抗(即具有各种癌症类型和治疗方案的CTR-DB数据集)之间的相关性,可以帮助确定该基因的功能重要性(图4B)。
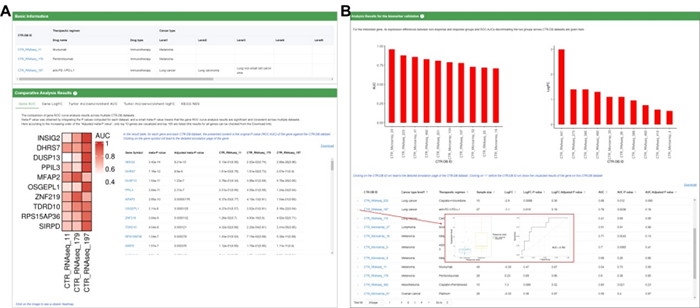
图4 数据集比较分析结果页面和标志物验证结果页面
CTR-DB对全球用户免费开放。未来,研究团队将会对CTR-DB进行持续维护和定期更新,将不断收录更多数据集,开发更多数据分析功能。
国家蛋白质科学中心李栋研究员、贺福初院士和刘中扬副研究员为论文共同通讯作者,刘中扬副研究员、刘佳乐硕士、刘欣悦硕士研究生为共同第一作者。
另外,该团队在过去五年研发了中药/小分子药物靶标预测和分析平台BATMAN-TCM(http://bionet.ncpsb.org.cn/batman-tcm/)、药物ATC分类预测器SPACE(http://lidong.ncpsb.org.cn/space/)等系列药物分析软件,其中BATMAN-TCM在过去五年为国内外近4万用户提供了超24万次的数据分析服务,被引300次(Google Scholar),被汤森路透公司的基本科学指标数据库(Essential Science Indicators)评为领域高被引论文。
原文链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab860/6376024
数据库链接:
http://ctrdb.ncpsb.org.cn
参考文献
[1] Schwaederle,M., Zhao,M., Lee,J.J., Eggermont,A.M., Schilsky,R.L., Mendelsohn,J., Lazar,V. and Kurzrock,R. (2015) Impact of precision medicine in diverse cancers: a meta-analysis of phase II clinical trials. J. Clin. Oncol., 33, 3817–3825.













