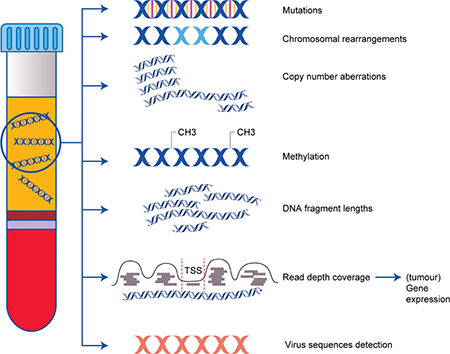
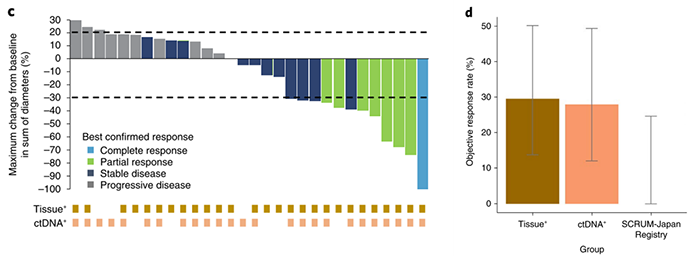
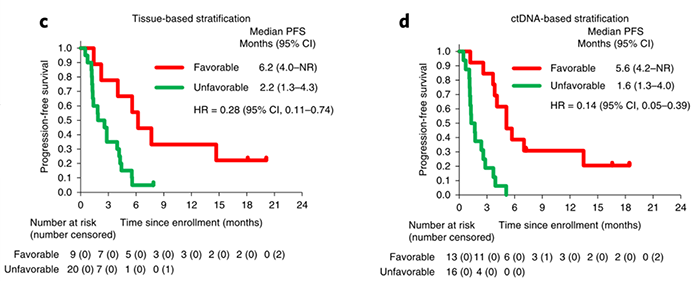
循环肿瘤DNA(circulating tumor DNA,ctDNA)是由肿瘤或循环肿瘤细胞(CTC)释放的游离DNA,存在于患者血浆中,长度通常少于200个核苷酸。ctDNA可能来自于肿瘤细胞的凋亡和坏死,也可能来自于活肿瘤细胞的主动释放,确切机制尚不清楚。使用ctDNA进行早期癌症检测可以获得与肿瘤相关的临床信息【1, 2】。这一方面是因为ctDNA可能直接反映肿瘤的基因组,另一方面是因为ctDNA的拷贝数与肿瘤类型、位置以及癌症分期都密切相关。从ctDNA可以获得许多与肿瘤相关的特征信息,诸如体细胞基因组畸变(突变、染色体重排、拷贝数改变)和表观遗传学畸变等(图1)。因此,使用ctDNA分析肿瘤细胞的基因组可以用于肿瘤的非侵入性检测和诊断,也可以帮助指导肿瘤的特异性治疗,同时能够监测整个治疗方案中的肿瘤进展。图1 ctDNA的不同特征及潜在的临床意义【2】。ctDNA可以作为生物标志物评估肿瘤情况,它的特异性好、准确度高,有助于明确肿瘤基因分型。已有研究评估了ctDNA基因分型在晚期胃肠道癌中的效用,与组织基因分型相比,ctDNA基因分型显著缩短了筛选时间(11 days vs 33 days,p<0.0001),提高了试验入组率(9.5% vs 4.1%,p<0.0001)【3】,并且没有降低治疗功效。ctDNA通常用于检测晚期实体肿瘤患者的基因组改变,但尚未确定在临床实践中对癌症患者实施ctDNA基因分型的适用性。近日,日本国立癌症研究中心东医院Takayuki Yoshino团队在Nature Medicine上发表了题为Circulating tumor DNA-guided treatment with pertuzumab plus trastuzumab for HER2 -amplified metastatic colorectal cancer : a phase 2 trial的研究论文,这是一项由研究者发起的名为TRIUMPH的二期临床试验,入组的是经ctDNA或肿瘤组织基因分型前瞻性确认的HER2阳性结直肠癌患者,评估帕妥珠单抗和曲妥珠单抗联合治疗的有效性和安全性。该临床试验达到了主要终点:阳性患者中确认的客观反应率约30%,联合治疗可能具有更好的疗效。更重要的是,无论是通过ctDNA基因分型还是传统的组织基因分型确认HER2阳性的mCRC患者,联合治疗都表现出相似的疗效,即ctDNA基因分型的准确性与组织基因分型相似。该临床试验也首次表明,经过ctDNA基因分型前瞻性确认的HER2阳性mCRC患者能够受益于双重HER2阻断治疗。TRIUMPH是一项在日本进行的多中心、非盲、单臂的II期临床试验。入组受试者为经组织学确认(IHC和FISH筛查)或经ctDNA测序确认(血浆NGS分析)的mCRC患者,均为HER2阳性(IHC 3+)。入组30名患者,其中22名患者为组织学和ctDNA测序共同确认(tissue+ ctDNA+),5名仅为组织学确认(tissue+ ctDNA-),3名为ctDNA测序确认(tissue- ctDNA+)。最终分别将 27 名和 25 名患者分别分配到tissue+和 ctDNA+ 组,每 3 周静脉注射帕妥珠单抗(840 mg负荷剂量,后续420 mg)和曲妥珠单抗(8 mg/kg负荷剂量,后续6 mg/kg)。TRIUMPH临床试验达到了主要终点(图2)。tissue+组27名患者中有8名(30%)达到了经确认的客观反应(95%置信区间(CI),14-50%),包括 1 名患者完全反应;ctDNA+组25名患者中有7名达到经确认的客观反应(95% CI,12-49%),包括1名患者完全反应。两组客观反应患者数目均超过拒绝零假设所需的5名。图2 帕妥珠单抗联合曲妥珠单抗治疗HER2阳性mCRC的临床疗效临床试验中tissue+组和ctDNA+组的临床疗效相似。tissue+组和ctDNA+组的中位无进展生存期(PFS)分别为4.0个月(95% CI,1.4-5.6)和3.1个月(95% CI,1.4-5.6),中位总生存期(OS)分别为10.1个月(95% CI,4.5-16.5)和8.8个月(95% CI,4.3-12.9)。此外,有24名患者(80%)发生了与治疗相关的不良事件(输液相关反应、腹泻、口腔炎和不适等),3名(10%)患者发生了3级治疗相关的不良事件,没有发生意外或治疗相关的死亡事件。临床试验后,研究者们进行了事后探索性分析,研究组织基因分型或ctDNA基因分型与临床疗效之间的关系。在来自无反应者的基线样本中,组织基因分型中RTK/RAS/PI3K相关基因的并发改变仅为19%(4/21),而ctDNA基因分型为 67%(14/21)(P = 0.004)。相关基因的并发突变与乳腺癌和胃癌HER2靶向治疗的耐药机制有关,并发突变显著富含在无反应患者中,意味着ctDNA基因分型能够较为准确地预测无反应者。接着,研究者们评估了肿瘤组织和ctDNA中的基线HER2状态与临床获益之间的关联。在基于组织基因分型中,相比利用FISH确定HER2/CEP17比率或HER2拷贝数,NGS确定HER2拷贝数与临床获益的相关性较为显著。在基于ctDNA分型中,利用肿瘤分数调整血浆ctDNA拷贝数(tumor-fraction-adjusted plasma CN,ApCN),基于ApCN的ctDNA分型与临床获益的相关性也一样显著。进一步,研究者们比较组织基因分型(基于NGS确定HER2拷贝数)和ctDNA分型(基于ApCN)在患者生存结果上的准确性(图3)。在组织基因分型方式中,HER2拷贝数 ≥ 68.7且无并发的RTK/RAS/PI3K突变定义为有望获益的患者(Favorable),反之定义为Unfavorable;在ctDNA基因分型方式中,ApCN中HER2拷贝数 ≥ 16.7且无并发的RTK/RAS/PI3K突变定义为有望获益的患者(Favorable),反之定义为Unfavorable。从结果中可以看到,没有致癌基因并发突变且HER2拷贝数高于阈值的患者,都显示出明显更好的无进展生存期(PFS)。并且,基于ctDNA的分型相比组织基因分型HR更低。这些发现表明基线ctDNA基因分型可以更准确地识别未来将从帕妥珠单抗与曲妥珠单抗联合治疗中受益的患者。图3 基于ctDNA和肿瘤组织基因分型后患者的生存结果总而言之,TRIUMPH临床试验达到了主要终点:帕妥珠单抗和曲妥珠单抗联合治疗在HER2扩增 mCRC的患者中展现了很好的有效性,阳性患者中的客观反应率约30%。事后探索性分析显示,基于HER2拷贝数的基线 ctDNA 基因对患者分型,其准确性与组织基因分型相似,这意味着ctDNA 基因分型可以识别受益于双重 HER2 阻断的患者并监测治疗反应。在临床实践中对mCRC患者实施ctDNA基因分型和组织HER2检测,有利于确定帕妥珠单抗和曲妥珠单抗双重HER2阻断的候选者。https://www.nature.com/articles/s41591-021-01553-w1. Nakamura, Y. & Shitara, K. Development of circulating tumour DNA analysis for gastrointestinal cancers. ESMO Open 5, (2020).2. Keller, L., Belloum, Y., Wikman, H. & Pantel, K. Clinical relevance of blood-based ctDNA analysis: mutation detection and beyond. Br. J. Cancer 124, 345–358 (2021).3. Nakamura, Y. et al. Clinical utility of circulating tumor DNA sequencing in advanced gastrointestinal cancer: SCRUM-Japan GI-SCREEN and GOZILA studies. Nat. Med. 26, 1859–1864 (2020).