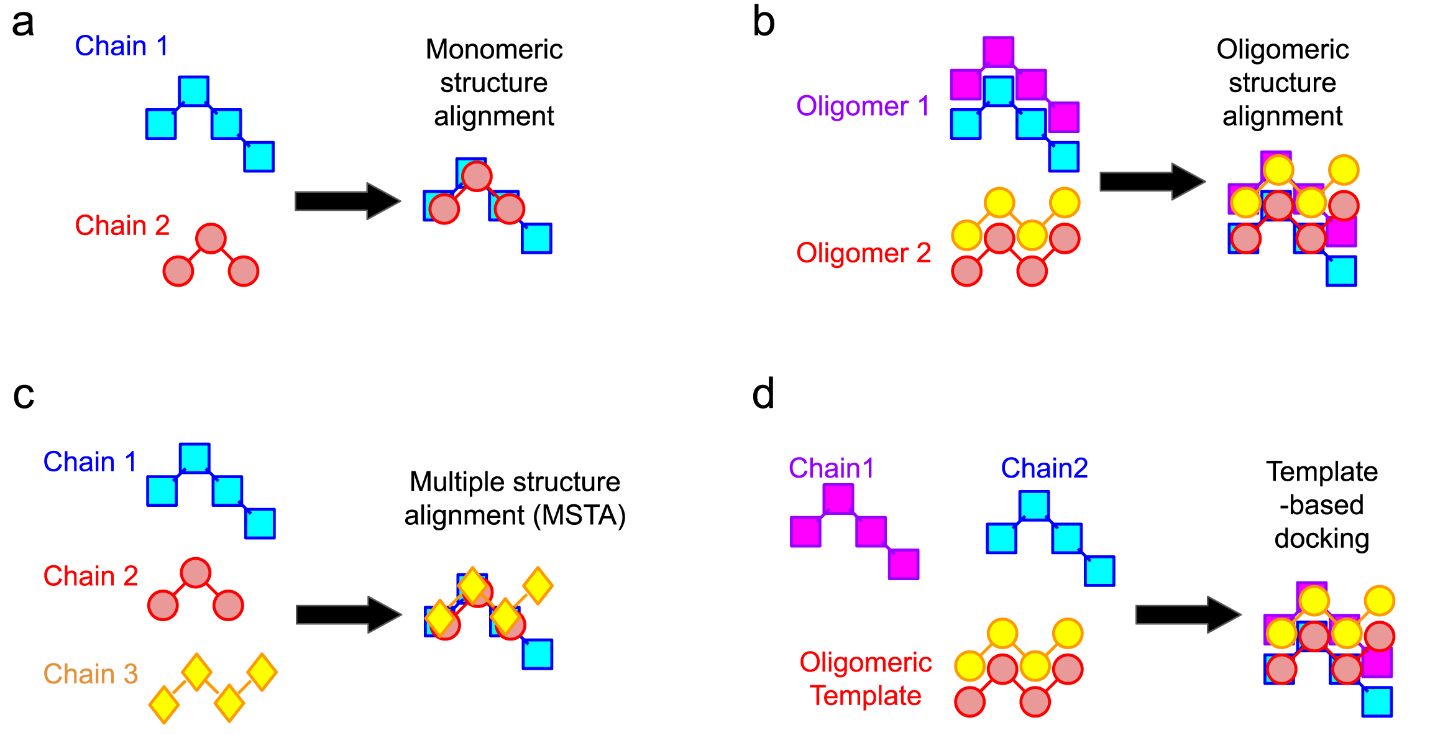
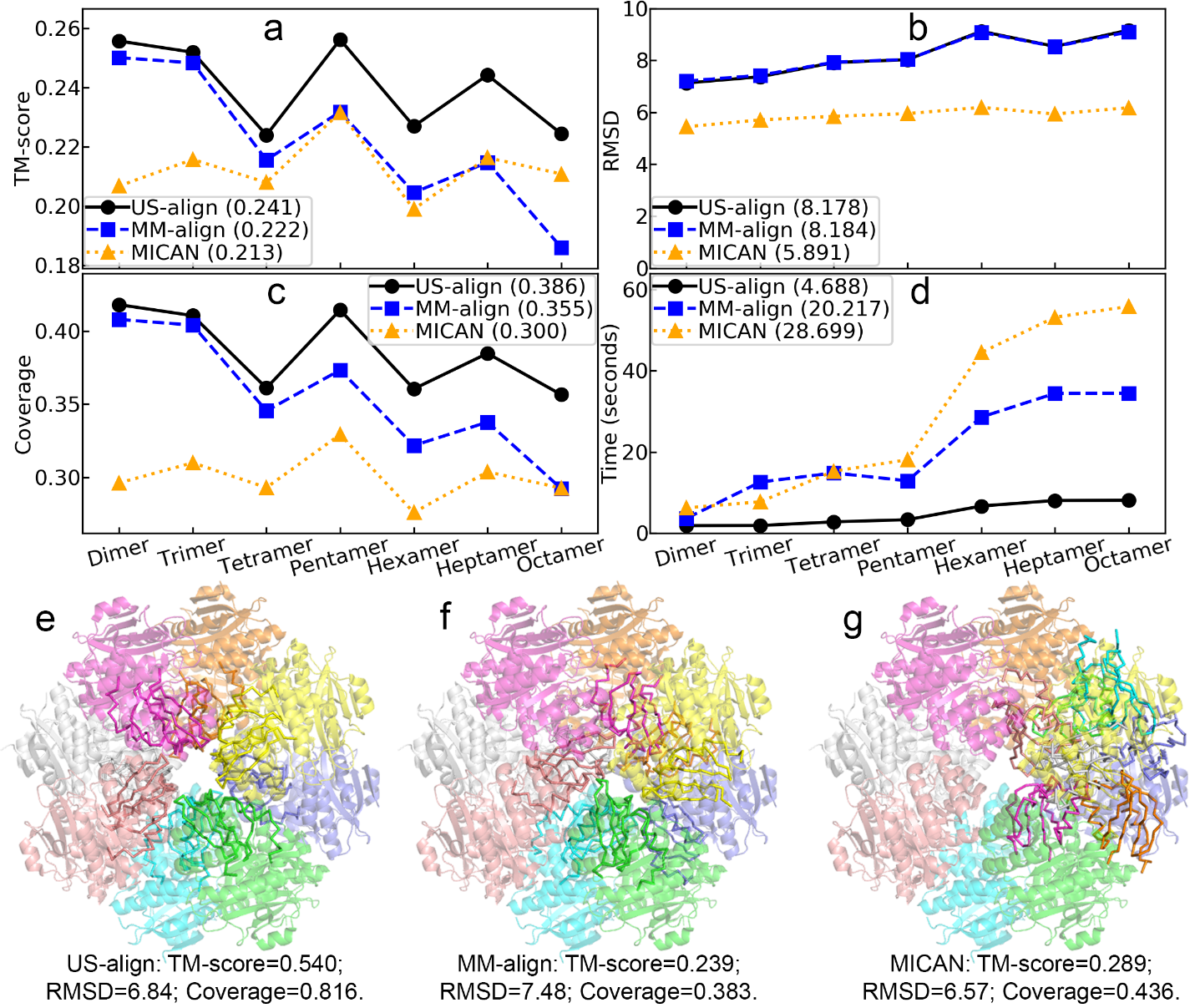
生物大分子的三维结构的比对算法在结构生物学和生物信息学中有着广泛的应用,包括结构分类、功能注释、药物发现和蛋白设计。密歇根大学张阳课题组多年来开发了多款高效准确的结构比对算法,包括用于蛋白比对的TM-align、用于RNA比对的RNA-align、用于蛋白复合物结构比对的MM-align等。无论是课题组开发的TM-align、RNA-align和MM-align,还是其它组开发的结构比对程序,都只是对特定的分子类型和特定的结构比对任务进行优化。这不但要求用户同时安装和使用多款不同的程序,也使得不同类型的大分子及其复合物之间的结构与功能比对变得困难。针对上述这些问题,2022年8月29日,密歇根大学张阳课题组张成辛博士等在Nature Methods上发表了文章US-align: Universal Structure Alignment of Proteins, Nucleic Acids and Macromolecular Complexes,开发了新一代比对算法US-align。US-align是第一款同时提供蛋白、RNA、DNA的单体及其复合物的两两比对以及多重比对的程序(图1),并且所有比对功能都统一使用的TM-score作为打分函数。US-align高度优化的C++代码使其在比其它在特定的结构比对任务上优化的程序的速度提高了数倍,并有更准确的结构比对结果。其中,蛋白与核酸的单链比对基本上分别沿用TM-align与RNA-align算法,多重比对借鉴mTM-align算法并新增RNA多重比对功能,而复合物结构比对算法则是完全从头开发,在准确性和速度上都超越旧版的MM-align(图2)。
图1. US-align所包含的结构比对功能。a, 单链结构的两两比对。b, 复合物结构的两两比对。c, 多重结构比对。d, 基于模板的分子对接。
图2. US-align与已有的复合物结构比对算法(MM-align和MICAN)在a, TM-score、b, RMSD、c, 覆盖率和d, 运行时间四方面的比较。e, US-align、f, MM-align与g, MICAN对两个八聚体复合物比对的结果,两个复合物PDB编号4JHM和4IAJ,分别由半透明卡通和实线彩带表示,不同颜色表示不同链。
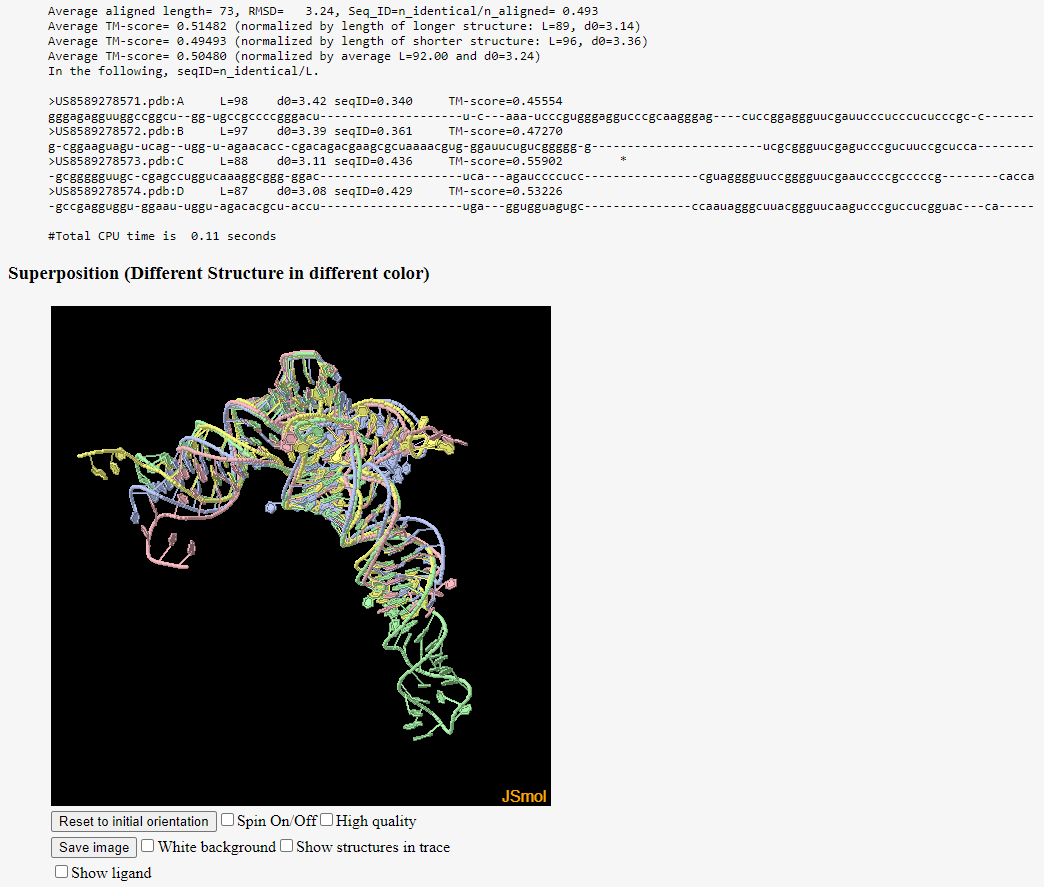
US-align是开源程序(https://github.com/pylelab/USalign),提供网页服务器以及单机版命令行程序下载,在Windows、Linux和Mac OS操作系统下都能流畅运行(图3)。新一版的US-align2的开发正在进行,将在保留所有旧版功能的基础上,添加非序列比对(Non-Sequential Alignment)等功能。
图3. US-align网页版(https://zhanggroup.org/US-align/)的多重结构比对结果。
原文链接:
https://doi.org/10.1038/s41592-022-01585-1